scDiffEq:使用神经随机微分方程对单细胞动态进行漂移扩散建模
原标题:scDiffEq: drift-diffusion modeling of single-cell dynamics with neural stochastic differential equations
5 分
关键词
摘要
单细胞测序测量通过捕捉单个细胞的快照分子特征,有助于重建动态生物学。在发育和疾病中,细胞命运决策是通过确定性和随机性调控事件的复杂平衡来协调的。漂移扩散方程在从高维单细胞测量中建模单细胞动态方面非常有效。现有的解决方案在细胞状态层面描述了与这些方程的漂移项相关的确定性动态,而扩散则被建模为跨细胞状态的常数。为了全面理解发育和疾病中的动态调控逻辑,需要明确调整确定性和随机性生物学平衡的模型。为了解决这些限制,我们引入了scDiffEq,这是一种用于学习神经随机微分方程的生成框架,可以近似生物学的确定性和随机性动态。使用谱系追踪的单细胞数据,我们证明了scDiffEq在重建细胞轨迹和预测造血过程中多能祖细胞的细胞命运方面提供了改进。通过对多能祖细胞进行计算机模拟扰动,我们发现scDiffEq准确地再现了CRISPR扰动的造血动态。我们将这种方法推广到超越谱系追踪或多时间点数据集,以建模来自单一时间点的单细胞数据的动态。使用scDiffEq,我们模拟了高分辨率的发育细胞轨迹,可以对其漂移和扩散进行建模,使我们能够研究其时间依赖的基因水平动态。
AI理解论文




该文档主要探讨了单细胞转录组数据中细胞动态的建模方法,特别是通过漂移扩散方程来模拟细胞状态的变化。文中介绍了如何利用这些方法来推断细胞的过去和未来状态,以及细胞状态与命运之间的关系。这些方法为研究发育过程和疾病中的调控动态提供了新的见解。
首先,文档描述了数据预处理的过程,采用了PRESCIENT的方法,过滤掉非高变异基因,并回归与细胞周期相关的基因。然后对对数归一化的表达计数进行缩放,并使用PCA(主成分分析)进行降维。这些步骤确保了数据的质量和可用性,为后续的建模提供了基础。
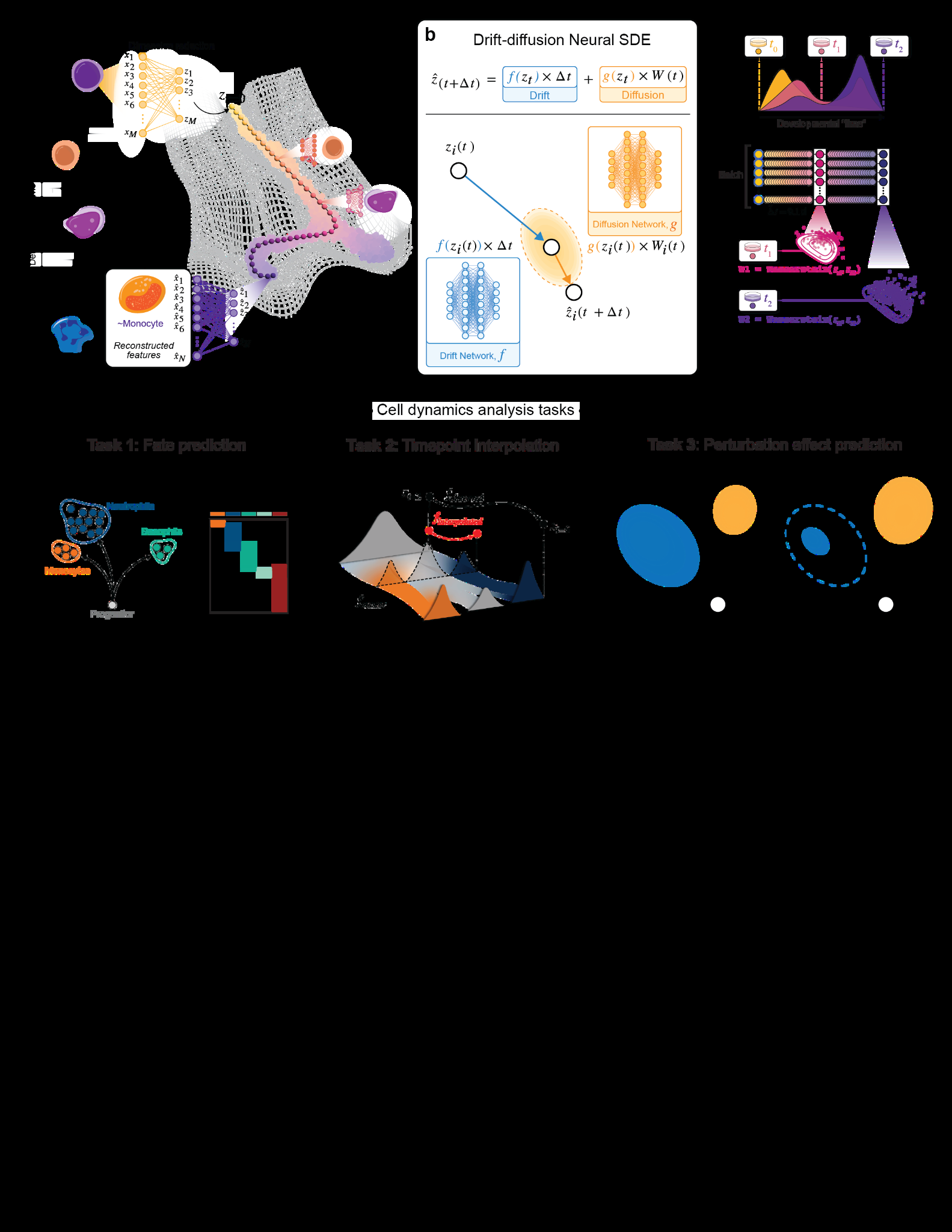
接下来,文档详细介绍了漂移扩散模型。该模型假设细胞状态的变化可以用漂移扩散方程表示,其中漂移项代表细胞状态的确定性变化,而扩散项则表示随机变化。通过引入潜在时间和布朗运动,模型能够模拟细胞在转录空间中的动态变化。为了约束漂移项,采用了Yeo等人的方法,将其定义为某个势函数的负梯度。这种方法允许通过时间积分来预测细胞的未来状态。
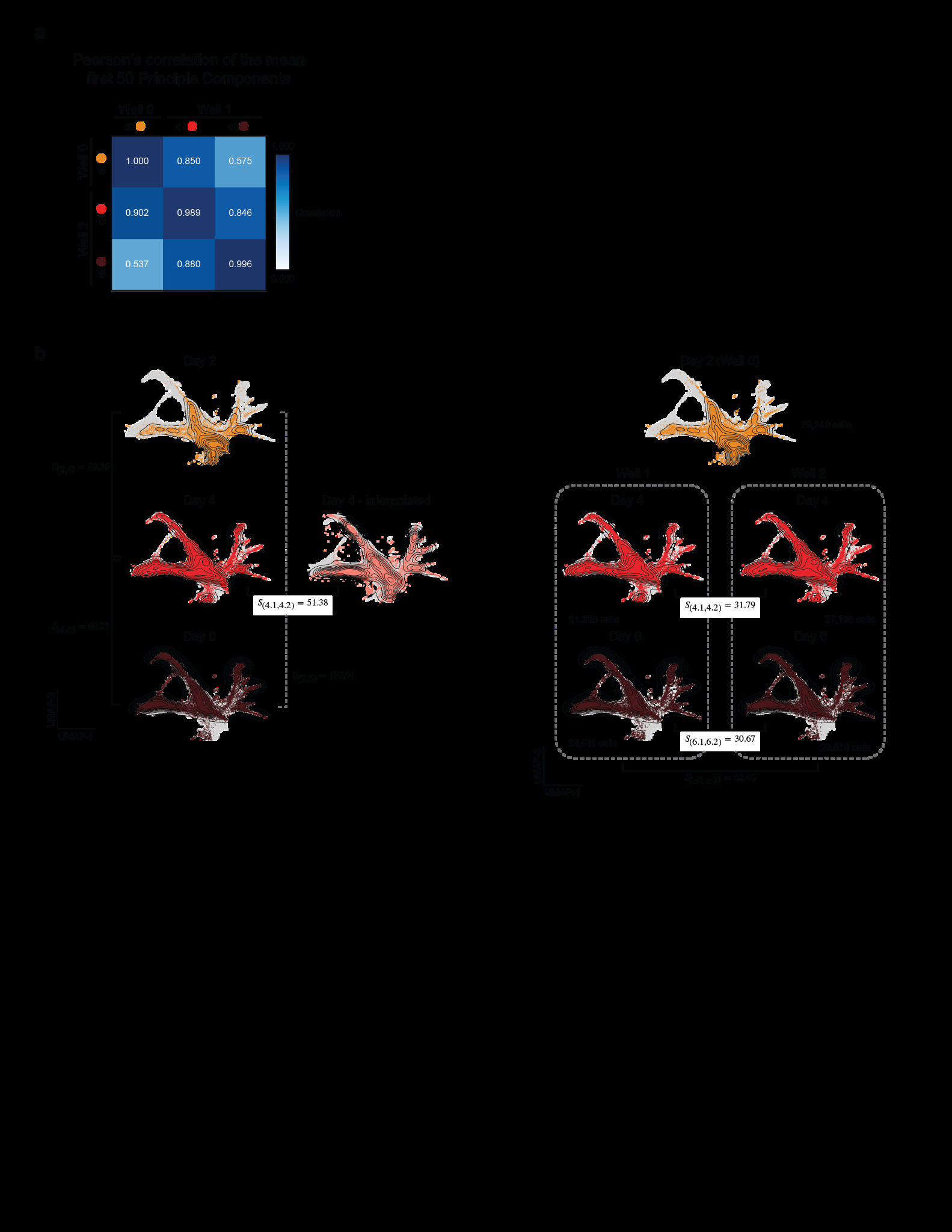
文档还介绍了Sinkhorn散度,这是一种用于比较模拟细胞群体与观察到的细胞群体的度量。Sinkhorn散度是一种无偏的熵正则化Wasserstein距离,能够有效地衡量两组细胞之间的差异。这种度量在模型的拟合过程中起到了关键作用,帮助评估模型的准确性。
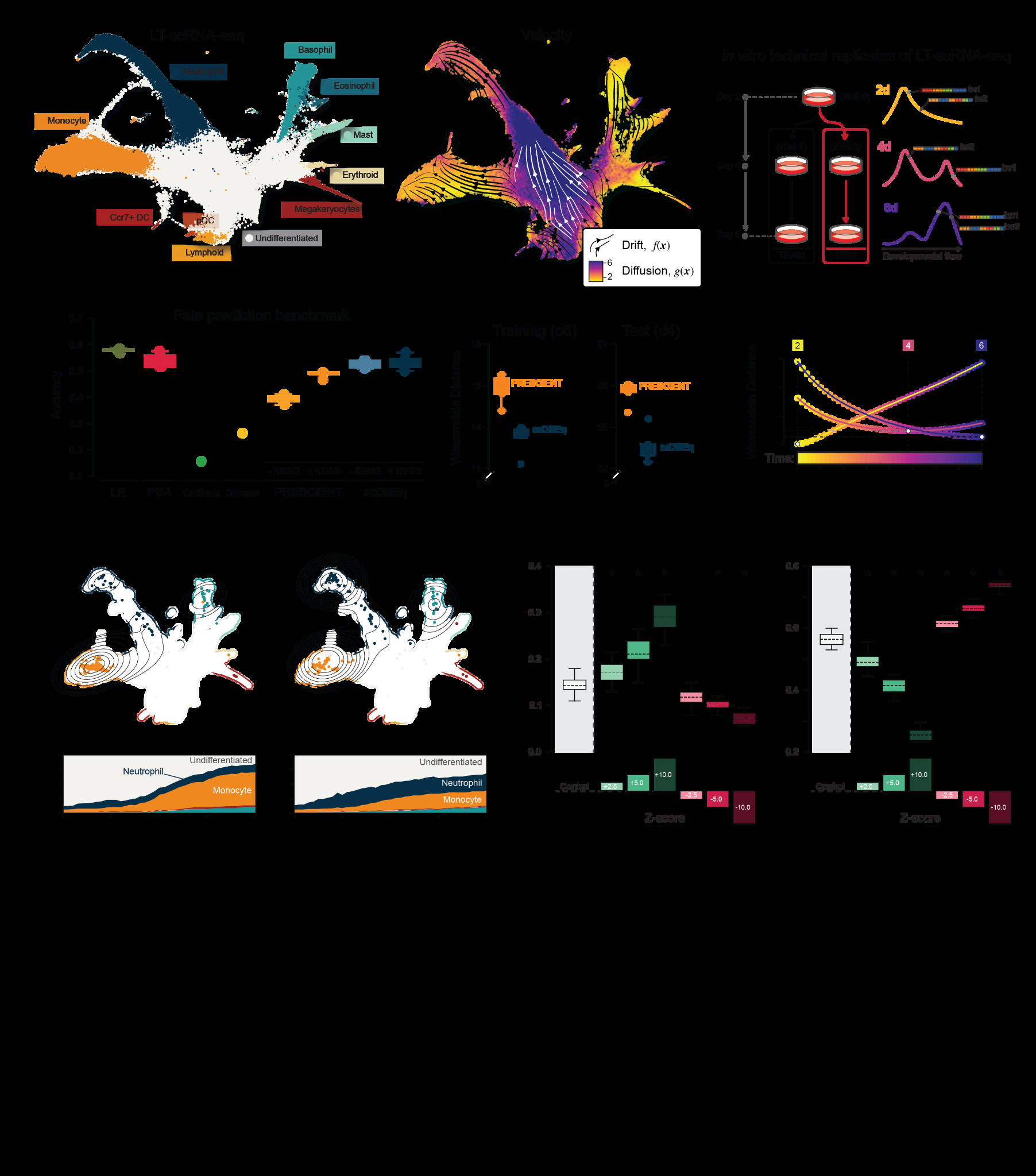
在方法的应用方面,文档提到了一些基于RNA速度的推断方法,如Dynamo和CellRank,这些方法利用RNA速度来推断细胞轨迹和命运。然而,这些方法对预处理和平滑操作非常敏感,难以准确建模多命运轨迹。相比之下,漂移扩散方程提供了一种更为稳健的框架,能够在高维空间中模拟细胞动态。
文档还提到了一些基准任务,用于评估模型在学习和预测细胞动态方面的准确性。这些任务包括命运偏向预测和未观察时间点的中间细胞状态预测。在命运预测任务中,文档比较了scDiffEq与其他方法的表现,发现分类器方法(如逻辑回归)在预测细胞命运方面表现更好,但这些方法在揭示分子过程和动态方面的信息有限。
最后,文档总结了这些方法在单细胞转录组数据分析中的应用潜力,强调了它们在揭示细胞状态和命运之间关系方面的重要性。这些方法不仅提供了对细胞动态的深刻理解,还为未来的研究提供了新的工具和视角。通过结合机器学习和生物学领域的知识,这些方法为研究细胞发育和疾病中的复杂动态过程提供了强有力的支持。
