GFETM:基于基因组基础的嵌入主题模型用于scATAC-seq建模
原标题:GFETM: Genome Foundation-based Embedded Topic Model for scATAC-seq Modeling
5 分
关键词
摘要
单细胞转座酶可及染色质测序(scATAC-seq)已成为研究单细胞分辨率开放染色质景观的强大技术。然而,由于数据的稀疏性和噪声,分析scATAC-seq数据仍然具有挑战性。基因组基础模型(GFMs)在大量DNA序列上进行预训练,已被证明在基因组分析中有效。鉴于开放染色质区域(OCRs)包含显著的序列特征,我们假设利用GFMs的序列嵌入可以提高scATAC-seq建模的准确性和泛化能力。在此,我们介绍了基因组基础嵌入主题模型(GFETM),这是一种可解释的深度学习框架,将GFMs与嵌入主题模型(ETM)结合用于scATAC-seq数据分析。通过整合从OCRs中提取的GFM DNA序列嵌入,GFETM展示了卓越的准确性和泛化能力,并通过零样本推理和注意力机制分析捕捉细胞状态特异的转录因子活性。最后,GFETM推断的主题混合揭示了肾脏糖尿病的生物学意义表观基因组特征。
AI理解论文
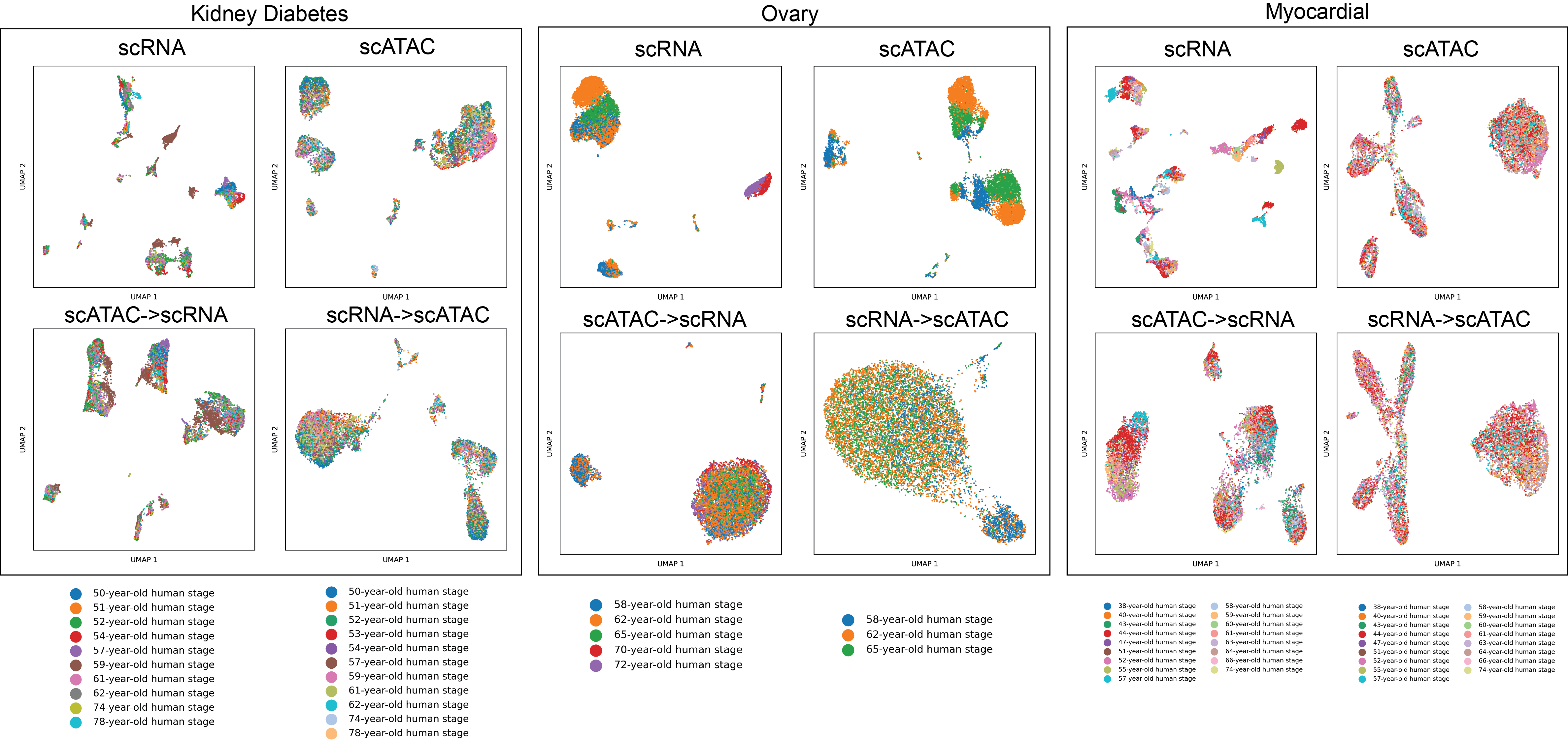
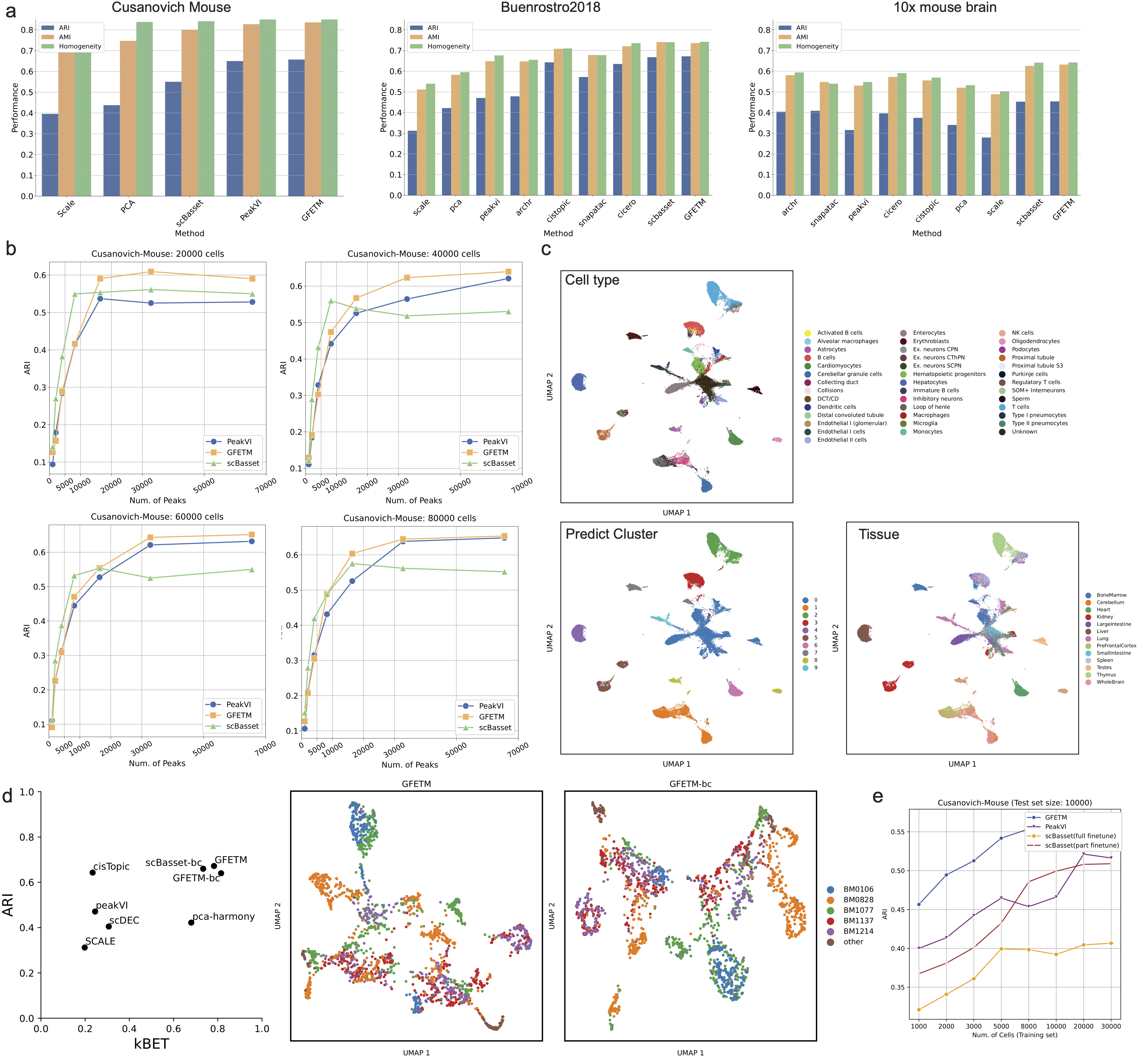



该文档主要介绍了一种名为**Genome Foundation Embedded Topic Model (GFETM)**的新型生成主题建模框架,该框架结合了基因组基础模型(GFMs)和嵌入式主题模型(ETM),用于分析单细胞ATAC-seq(scATAC-seq)数据。GFETM的设计旨在通过整合DNA序列信息来提高scATAC-seq建模的性能,并提供新的生物学见解。
背景与动机:单细胞ATAC-seq技术用于测量染色质可及性,帮助研究细胞的基因调控网络。然而,由于染色质区域远大于转录区域,信噪比较低,导致数据分析的复杂性。现有的计算方法分为两类:序列无关方法(如PCA、Cierco、SCALE、cisTopic)和序列相关方法(如chromVAR、BROCKMAN、scBasset)。序列无关方法忽略了DNA序列信息,可能导致性能不佳,而序列相关方法通过整合DNA序列信息改善了细胞嵌入,但在未见过的细胞上缺乏泛化能力。
GFETM框架:GFETM结合了ETM和GFM的优势。ETM是一种基于变分自编码器(VAE)的强大主题模型,已在单细胞RNA测序(scRNA-seq)建模和多组学整合中表现出色。GFETM通过联合训练ETM和GFM,将GFM生成的DNA序列嵌入用作ETM中的峰值嵌入。GFETM的设计允许GFM的序列知识无缝集成到ETM中,并通过线性解码器提供可解释性,从而增强整体性能。
方法与实现:GFETM的实现包括一个编码器和一个线性解码器,涵盖主题嵌入、峰值嵌入和批次效应截距。GFM接收峰值的DNA序列作为输入,生成序列嵌入,这些嵌入被用作ETM中的峰值嵌入。通过最大化变分下界(ELBO)进行训练,GFETM能够在不同实验、物种和组织生成的数据集之间进行零样本或少样本迁移学习。
实验与结果:GFETM在跨组织迁移学习中表现出色,特别是在细胞数量有限或质量较低的数据集上。通过对比不同组织间的零样本迁移性能,GFETM展示了其在不同组织间的优越性。此外,GFETM在处理大规模数据集时表现出良好的扩展性。
贡献与意义:GFETM的主要贡献在于其创新性地结合了GFM和ETM,提供了一种高效且可解释的scATAC-seq数据分析方法。其在跨数据集、跨物种和跨组织的迁移学习能力,特别是在处理低质量数据集时的表现,展示了其在生物信息学研究中的潜力。GFETM不仅提高了细胞嵌入的准确性,还为理解复杂的基因调控网络提供了新的视角。
术语解释:
- scATAC-seq:单细胞ATAC-seq技术,用于测量染色质的开放性。
- GFMs:基因组基础模型,通过大规模预训练生成DNA序列嵌入。
- ETM:嵌入式主题模型,一种基于变分自编码器的主题建模方法。
- VAE:变分自编码器,一种生成模型,用于学习数据的潜在表示。
- ELBO:变分下界,用于优化变分推断模型的目标函数。
通过GFETM的开发,研究人员能够更精确地分析和解码单细胞水平的染色质重塑过程,为生物学研究提供了强有力的工具。
