使用机器学习预测小分子的抗菌类别特异性
原标题:Predicting antimicrobial class specificity of small molecules using machine learning
5 分
关键词
摘要
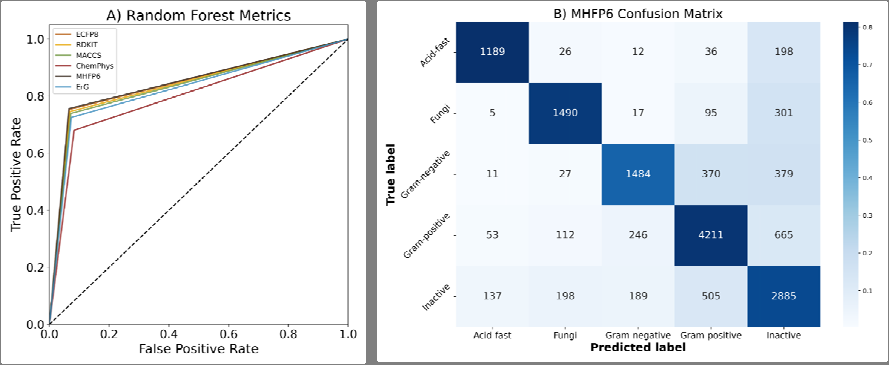
尽管由于耐药病原体的出现,抗生素药物的有效武器库不断减少,但新型治疗方法的开发也有所放缓。在先进计算方法的时代,机器学习(ML)等方法可能成为一种潜在的解决方案,以帮助降低抗生素药物发现的高成本和复杂性,并吸引跨组织的合作。在我们的工作中,我们开发了一个大型的抗菌知识图谱(AntiMicrobial-KG),作为收集和可视化公共体外抗菌实验的存储库。利用这些数据,我们构建了机器学习模型,以有效扫描化合物库,识别具有潜在抗菌活性的化合物。我们的策略涉及在六种化合物指纹表示中训练七种经典机器学习模型,其中基于MHFP6指纹训练的随机森林表现最佳,显示出75.9%的准确率和0.68的Cohen's Kappa分数。最后,我们展示了该模型在预测两个小分子筛选库的抗菌特性方面的适用性。首先,EU-OpenScreen库针对一组革兰氏阳性、革兰氏阴性和真菌病原体进行了测试。在这里,我们揭示了该模型能够正确预测超过30%的革兰氏阳性、革兰氏阴性和真菌病原体的活性化合物。其次,对于Enamine库,一个声称具有抗菌特性的商业可用HTS化合物集合,我们预测了其抗菌活性和病原体类别特异性。这些结果可能通过仔细筛选出商业库中活性较低的化合物,为加速抗菌药物耐药性药物发现的研究提供了一种手段。
AI理解论文



该文档主要探讨了通过机器学习(ML)模型预测化合物的抗菌活性,特别是针对不同病原体类别(如革兰氏阳性菌、革兰氏阴性菌、抗酸菌和真菌)的活性。研究的核心在于化合物指纹的使用,这些指纹是化合物结构特征的简化表示,用于捕捉化合物的物理化学性质、结构连接性和药效团内容。
首先,研究通过**合成少数类过采样技术(SMOTE)**解决了化合物在五个活性组中的不平衡问题,从而显著提高了模型预测的准确性。SMOTE是一种用于处理数据不平衡的技术,通过生成合成样本来平衡数据集中的类别分布。
其次,研究采用了系统的、渐进的选择标准来训练模型,以识别特定用例的最佳模型。研究比较了多种机器学习模型,发现随机森林模型在性能上优于其他模型,如朴素贝叶斯模型。通过这种比较,研究确定了最佳的模型-指纹组合,即MHFP6-随机森林组合,在所有可能的组合中表现最佳。
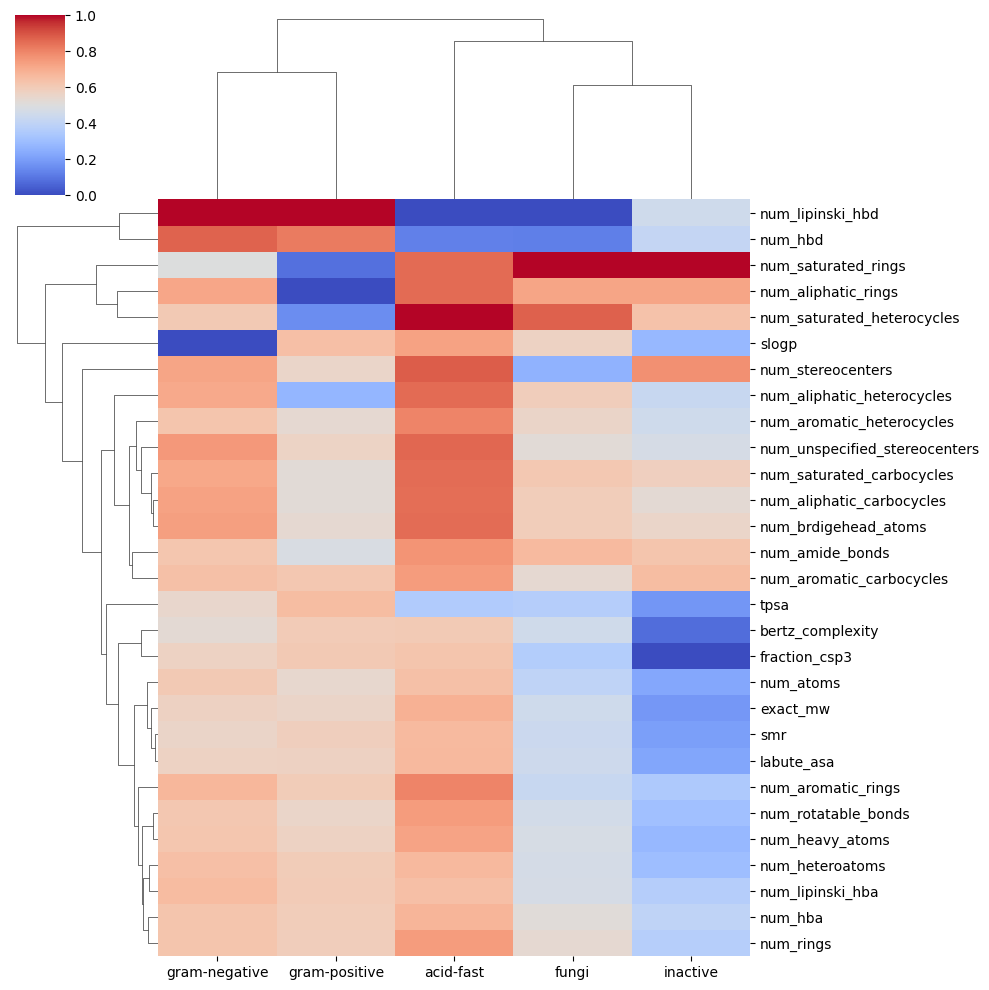
此外,研究还强调了经典结构和药效团指纹在性能上优于物理化学性质指纹。然而,基于化合物物理化学表示的模型训练可以为药物开发和优化提供有价值的见解。例如,研究发现氢键供体和LogP是影响抗真菌和抗菌活性的关键特征,这与现有研究一致。
研究还展示了模型预测在两个常用药物发现化合物库(EU-OPENSCREEN和Enamine抗菌集合)中的应用,旨在通过提高筛选选项来减少筛选成本,并帮助商业库供应商通过包括更多活性分子来增强其集合。
然而,研究也指出了当前方法的局限性。首先,化合物与病原体类别的关联基于最高记录的MIC活性,这可能导致误导,因为许多“广谱”化合物在多个病原体类别中表现出活性。此外,数据收集的异质性和模型验证集的结构多样性不足也限制了模型的泛化能力。
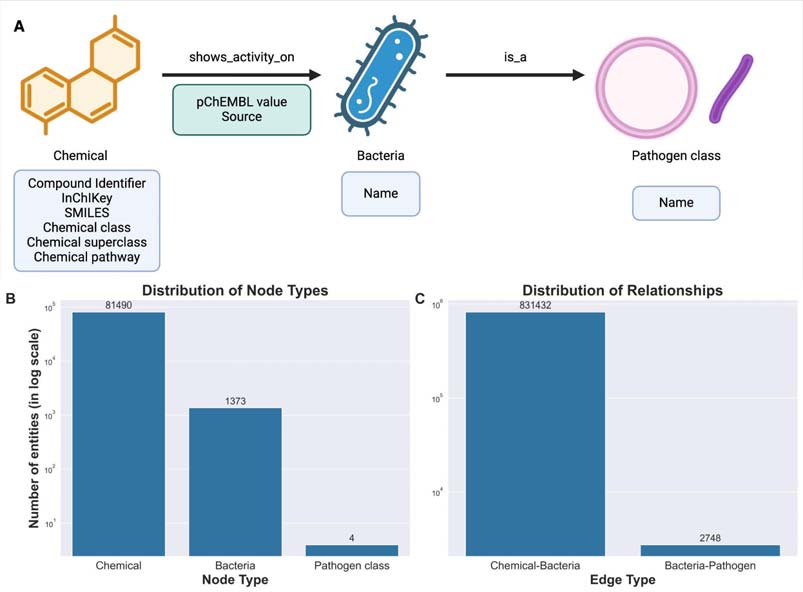
在方法方面,研究构建了一个名为**抗菌耐药知识图谱(AntiMicrobial-KG)**的数据仓库,整合了来自多个公共资源的实验验证抗菌化学品数据。该知识图谱采用属性图结构,支持复杂的查询和分析,促进对抗菌耐药性模式的深入理解。
研究通过将化学数据分为训练集和测试集来训练和评估机器学习模型。化合物首先根据其pChEMBL值分类为活性或非活性,并使用“最佳四分之一”方法确定病原体类别选择性。为了增强模型的鲁棒性,研究在数据集拆分期间采用了SMOTE技术,仅对训练数据应用,以确保测试集保持真实数据分布的准确表示。
最后,研究开发了一个可重复的管道来测试多个模型并优化表现最佳的模型。该管道利用PyCaret的模型比较功能,训练了六种经典机器学习模型,包括朴素贝叶斯(NB)、**逻辑回归(LR)和轻量梯度提升机(LightGBM)**等。
总之,该研究通过整合多种化合物指纹和机器学习技术,提供了一种有效的化合物活性预测方法,尽管存在一些局限性,但为未来的研究提供了坚实的基础。
