ActinoMation:一种用于中等通量链霉菌属机器人接合的文献化编程方法
原标题:ActinoMation: a literate programming approach for medium-throughput robotic conjugation of Streptomyces spp
5 分
关键词
摘要
链霉菌属是抗生素和其他药用重要生物活性化合物的宝贵生产者。分子工程工具的进步,如CRISPR,已经为链霉菌的代谢潜力提供了一些访问途径,但菌株的高效基因工程受到繁琐且缓慢的手动转化协议的阻碍。在本文中,我们提出了一种半自动化的中等通量工作流程,利用经济实惠且开源的Opentrons (OT-2)机器人平台将重组DNA引入链霉菌属。为了提高工作流程的可访问性,我们提供了一个开源协议创建器,ActinoMation。ActinoMation是一个在Jupyter Notebook中使用Python的文档化编程环境。我们通过将质粒pSETGUS和pIJ12551转化到链霉菌青霉素(M1152和M1146)、S. albidoflavus(J1047)和S. venezuelae(DSM40230)中验证了该方法。我们展示了M1152与pSETGUS和pIJ12551的结合效率为3.33*10−3;M1146与pSETGUS和pIJ12551的结合效率为2.96*10−3;J1047与pSETGUS的结合效率为1.21*10−5,与pIJ12551的结合效率为4.70*10−4,以及DSM40230与pSETGUS的结合效率为4.97*10−2,与pIJ12551的结合效率为6.13*10−2,误报率在8.33%到54.54%之间。结合工作流程的自动化在处理大样本量时提高了一致性,从而促进了更大规模的易于重复性。
AI理解论文



该文档主要介绍了一种自动化的中等通量工作流程,用于大肠杆菌(E. coli)与链霉菌(Streptomyces spp.)的基因工程。该流程结合了OT-2平台和Jupyter Notebook中的文字化编程,以实现实验的自动化和标准化。以下是该文档的主要内容和结构:
1. 背景与动机
文档首先介绍了基因工程在微生物研究中的重要性,特别是在链霉菌中,这类细菌以其生产次级代谢产物(如抗生素)而闻名。传统的基因工程方法通常耗时且易出错,因此需要一种高效、可重复的自动化流程来提高实验效率和准确性。
2. 方法与实现
2.1 自动化平台
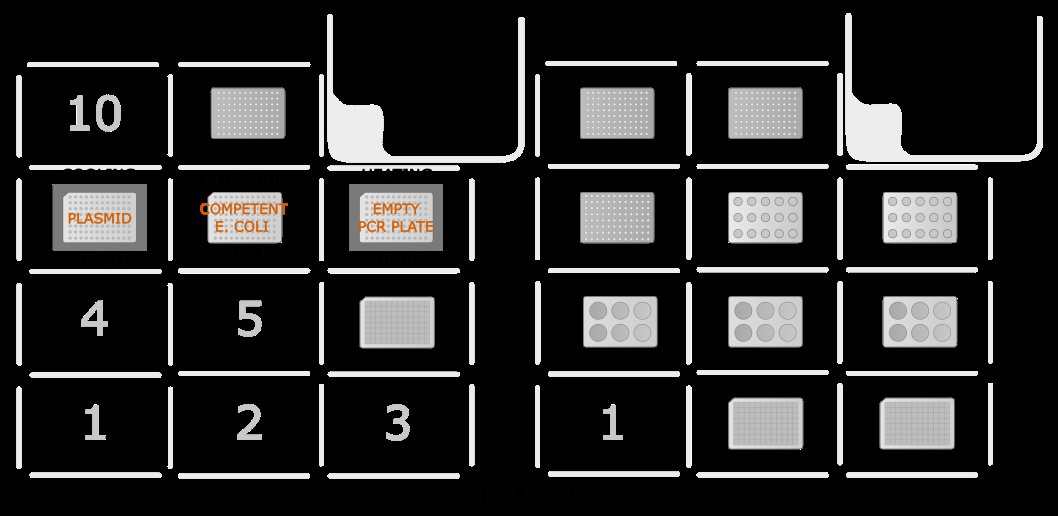
该研究使用了Opentrons OT-2机器人,这是一种开放源代码的液体处理机器人,能够执行复杂的实验操作。通过结合文字化编程,研究人员开发了一套用户友好的图形用户界面(GUI),使得非编程背景的用户也能轻松操作机器人。
2.2 文字化编程
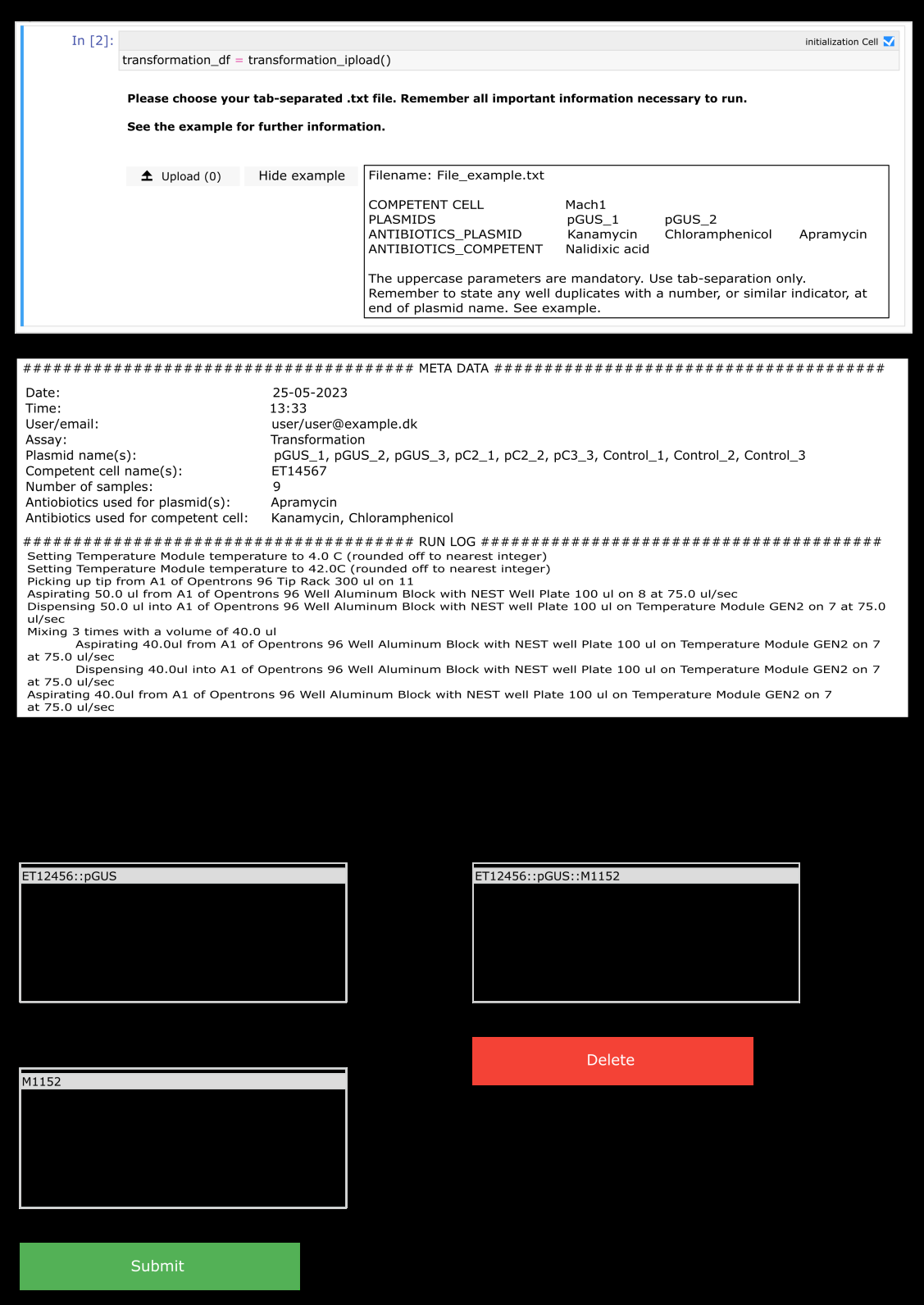
文字化编程是一种将自然语言描述转化为计算机代码的方法。在本研究中,使用了Jupyter Notebook来创建实验协议。用户通过GUI输入实验设置,系统自动生成Python协议、材料清单和运行日志。这种方法不仅提高了实验的可重复性,还减少了人为错误。
2.3 实验流程
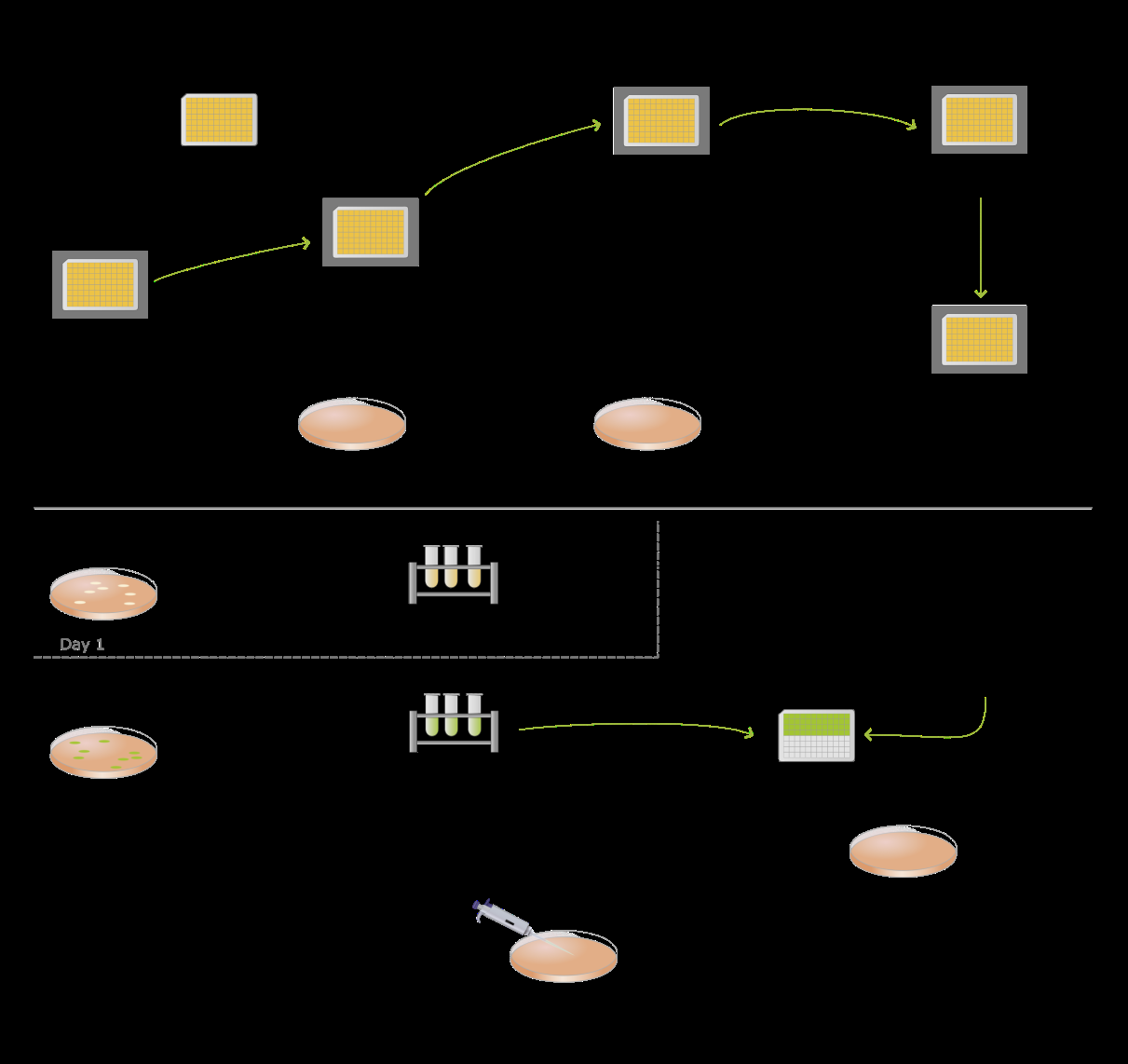
文档详细描述了热休克转化和链霉菌接合的实验流程。热休克转化涉及将质粒DNA引入大肠杆菌中,而接合则是将大肠杆菌中的质粒转移到链霉菌中。整个流程通过OT-2机器人自动化执行,确保了实验的一致性和高效性。
3. 实验验证
研究人员通过将两种质粒(pSETGUS和pIJ12551)转化到大肠杆菌中,并随后与链霉菌进行接合,验证了该系统的有效性。实验结果表明,该自动化流程能够成功实现基因转移,并且与手动操作相比,具有更高的效率和准确性。
4. 结果与讨论
4.1 成果
该研究的主要贡献在于开发了一种易于适应、成本效益高且可并行化的工作流程,适用于链霉菌的基因工程。通过结合自动化硬件和文字化编程,研究人员提供了一种标准化的实验设计和执行方法,减少了实验中的人为偏差。
4.2 挑战与局限
尽管该系统实现了许多实验步骤的自动化,但仍有一些步骤需要手动操作,如接合混合物的划线和抗生素覆盖层的添加。此外,由于链霉菌孢子的生长需要较大的生物量,某些实验步骤无法在小规模的96孔板上进行。
5. 未来展望
文档最后讨论了该系统的潜在应用和未来发展方向。研究人员鼓励其他用户在此基础上进行复制和开发,以促进透明度和跨学科合作。通过开放源代码的硬件和软件,其他研究团队可以采用这种设置,节省时间和成本。
术语解释
- 基因工程:通过人工手段改变生物体的遗传物质,以实现特定功能或特性。
- 链霉菌(Streptomyces spp.):一种土壤细菌,以生产抗生素和其他次级代谢产物而闻名。
- 文字化编程:一种编程范式,允许用户通过自然语言描述生成计算机代码。
- Opentrons OT-2:一种开放源代码的实验室自动化平台,用于液体处理和实验操作。
通过以上内容,读者可以全面理解该文档的研究背景、方法、实验验证及其在基因工程领域的贡献。
