基于结构的模拟的前瞻性评估显示其预测激酶突变对抑制剂结合影响的能力。
原标题:Prospective evaluation of structure-based simulations reveal their ability to predict the impact of kinase mutations on inhibitor binding
5 分
关键词
摘要
小分子激酶抑制剂是现代癌症治疗中至关重要的一部分,这一点从已有超过80种获得FDA批准的小分子激酶抑制剂可以看出。不幸的是,固有或获得的耐药性常常导致治疗中断,这通常是由于激酶治疗靶点的突变引起的。临床肿瘤测序的出现为精准肿瘤学提供了更多的机会,通过将最佳治疗与肿瘤突变谱相配对来改善患者的治疗效果。然而,现代精准肿瘤学的努力因缺乏足够的生化或临床证据来分别将每种突变分类为对现有抑制剂耐药或敏感而受到阻碍。基于结构的方法在预测激酶突变是否会影响抑制剂结合方面在回顾性基准测试中显示出很高的准确性,但这种比较是通过在不同条件下汇聚不同的实验测量结果进行的。我们展示了基于结构的方法在一个盲测数据集上首次进行的前瞻性基准测试,该数据集使用NanoBRET报告系统测量细胞内激酶抑制剂对Abl激酶突变体的亲和力。我们将NanoBRET结果与基于结构的方法进行比较以评估其对突变对抑制剂结合影响的估计(以ΔΔG度量)。通过比较基于物理的模拟、Rosetta和以前的机器学习模型,我们发现基于结构的方法能够准确分类激酶突变是否对抑制剂产生耐药性或增敏作用,各种方法都具有相似的准确度。我们显示,基于物理的模拟最适合估算远离激酶活性位点的突变的ΔΔG。为了探究失败模式,我们回顾性研究了两个临床上重要的但预测不佳的突变,T315A和L298F,发现起始构象和质子化状态显著影响我们预测的准确性。我们的实验和计算测量为评估未来方法和基于结构模型对突变对抑制剂结合亲和力影响提供了基准。这些基于结构的方法在识别肿瘤特异性突变的最佳治疗方案、预测临床数据缺乏时的耐药突变和识别对已建立抑制剂的潜在增敏突变方面具有潜在应用价值。
AI理解论文
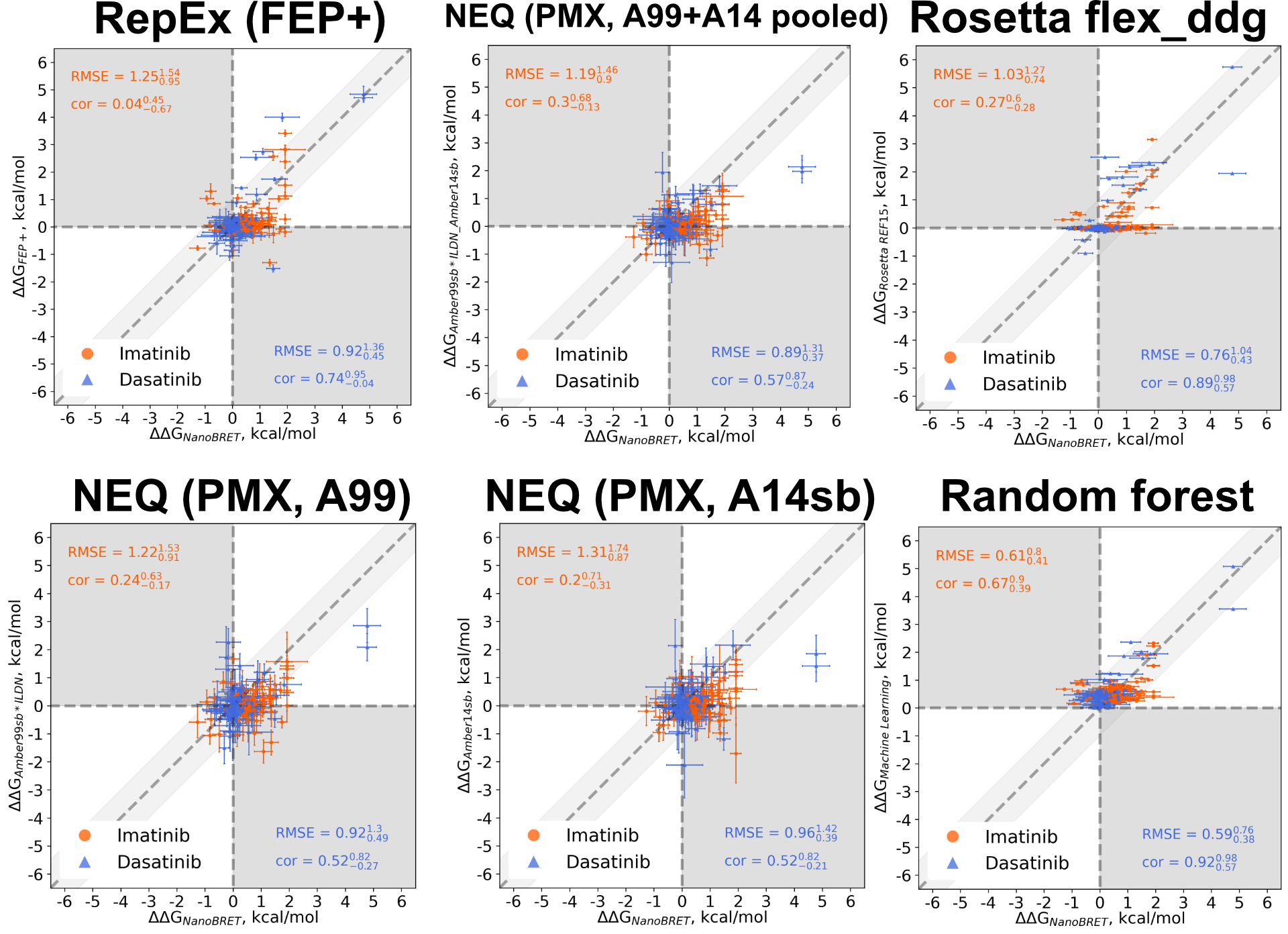


这篇论文探讨了小分子激酶抑制剂在癌症治疗中的重要性,尤其是在面对由激酶靶点突变引起的耐药性时的挑战。论文的核心是通过结构基础方法预测激酶突变对抑制剂结合的影响,并提供了一个前瞻性基准,用于评估这些方法在预测突变对抑制剂结合亲和力影响方面的能力。
引言
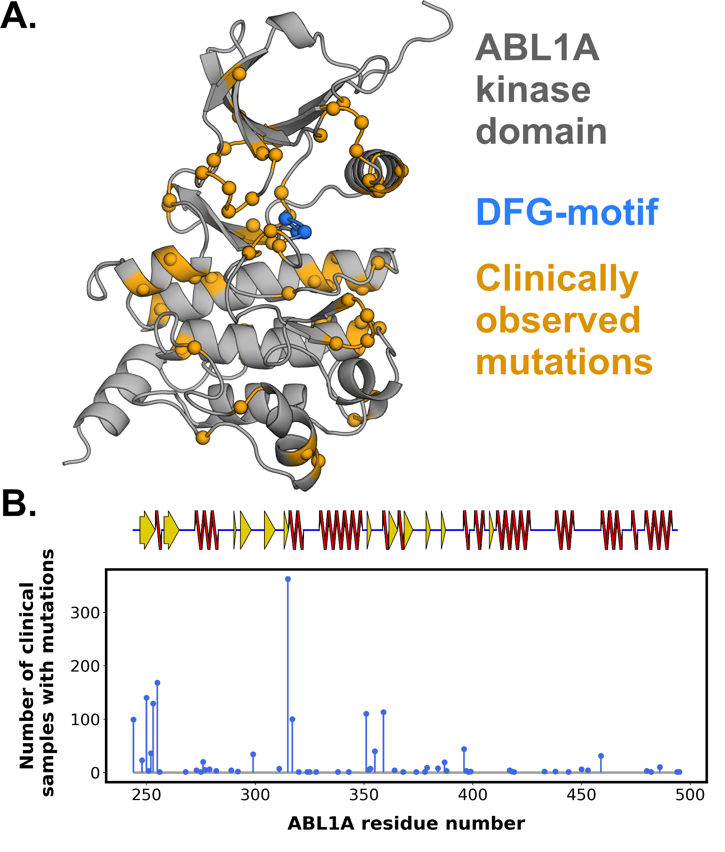
论文首先介绍了小分子激酶抑制剂在现代癌症治疗中的关键作用,指出超过80种此类抑制剂已获得FDA批准。然而,内在或获得性耐药性常常导致治疗中断,这通常是由激酶靶点的突变引起的。随着临床肿瘤测序的出现,精准肿瘤学有了新的机会,通过将最佳疗法与肿瘤突变谱配对来改善患者的治疗效果。然而,当前的精准肿瘤学努力受到缺乏足够的生化或临床证据的阻碍,无法将每个突变分类为对现有抑制剂的耐药或敏感。
方法
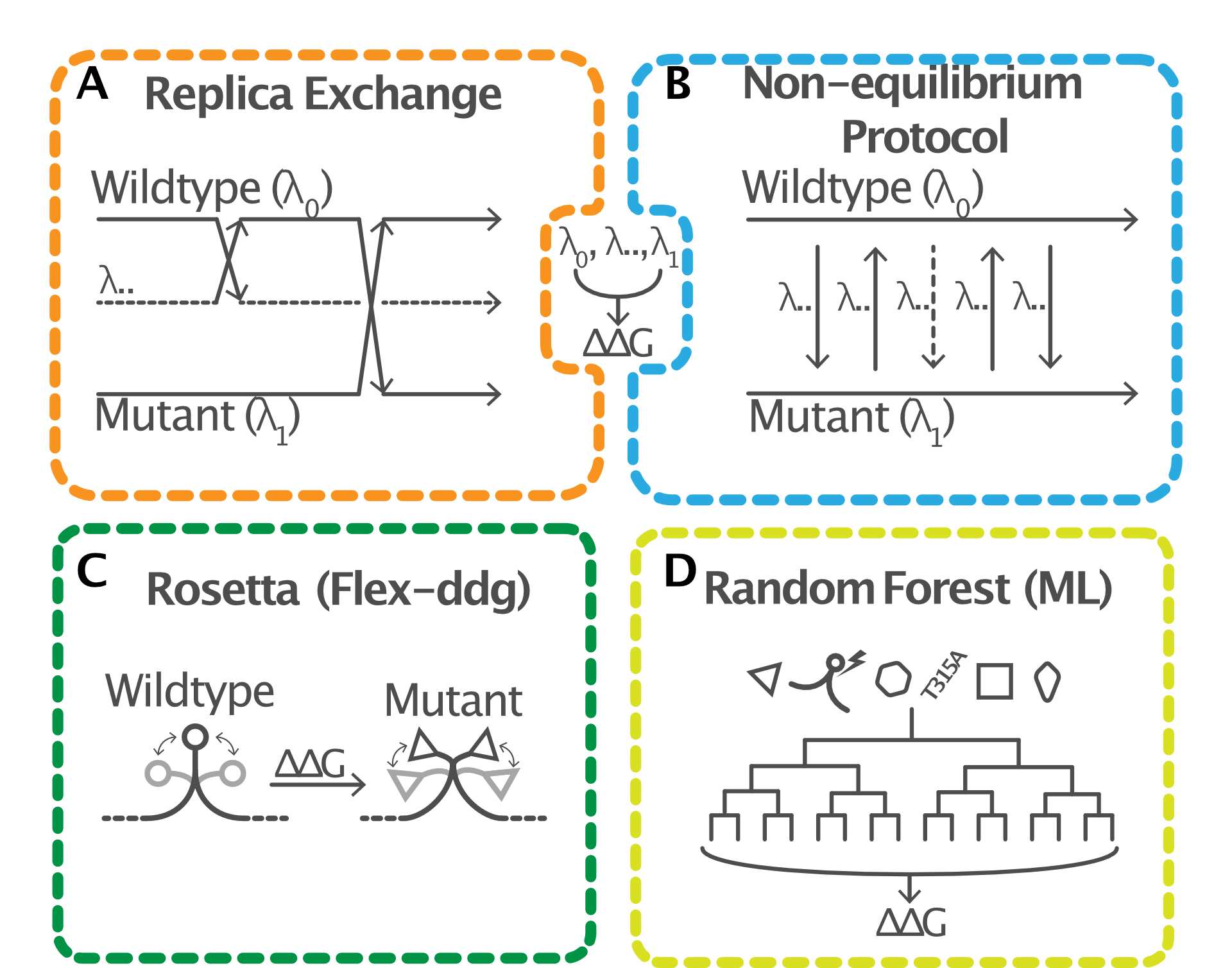
论文采用了结构基础方法,包括物理模拟、Rosetta和机器学习模型,来估计突变对抑制剂结合的影响(以ΔΔG衡量)。这些方法在一个盲测数据集上进行了测试,该数据集使用NanoBRET报告基因测定法测量了Abl激酶突变体的细胞内激酶抑制剂亲和力。
-
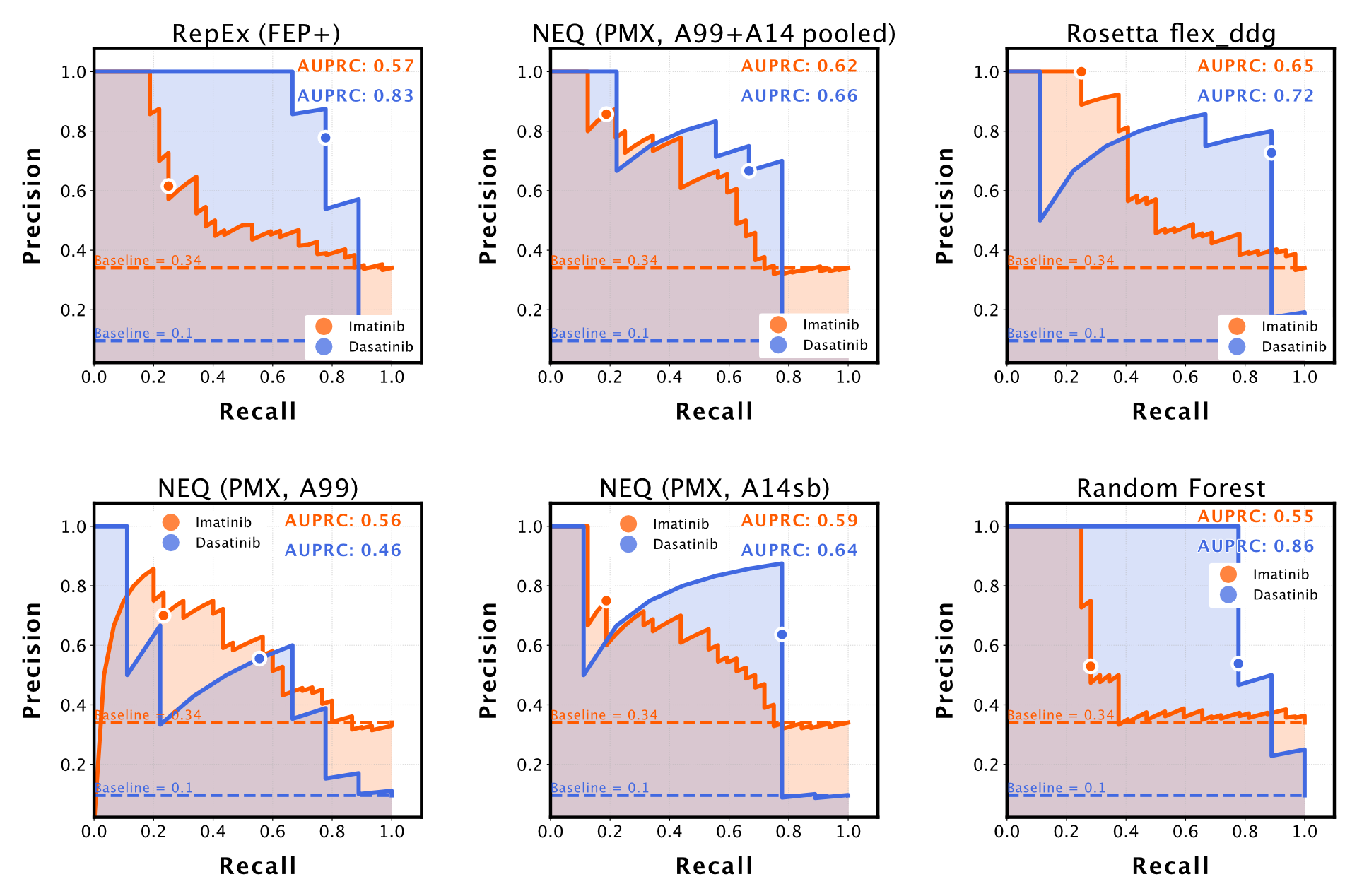
物理模拟:使用Gromacs进行分子动力学模拟,结合非平衡协议(NEQ)和PMX工具进行ΔΔG估计。模拟包括能量最小化、等温等压系综(NPT)下的平衡模拟,以及生产模拟。
-
Rosetta预测:使用flex_ddg协议计算结合自由能变化,采用REF2015和βNOV16评分函数。
-
机器学习模型:基于Python的ExtraTreesRegressor类构建,使用Hauser等人的数据集进行训练,数据集按突变进行分割以提高对新突变的外推性能。
实验
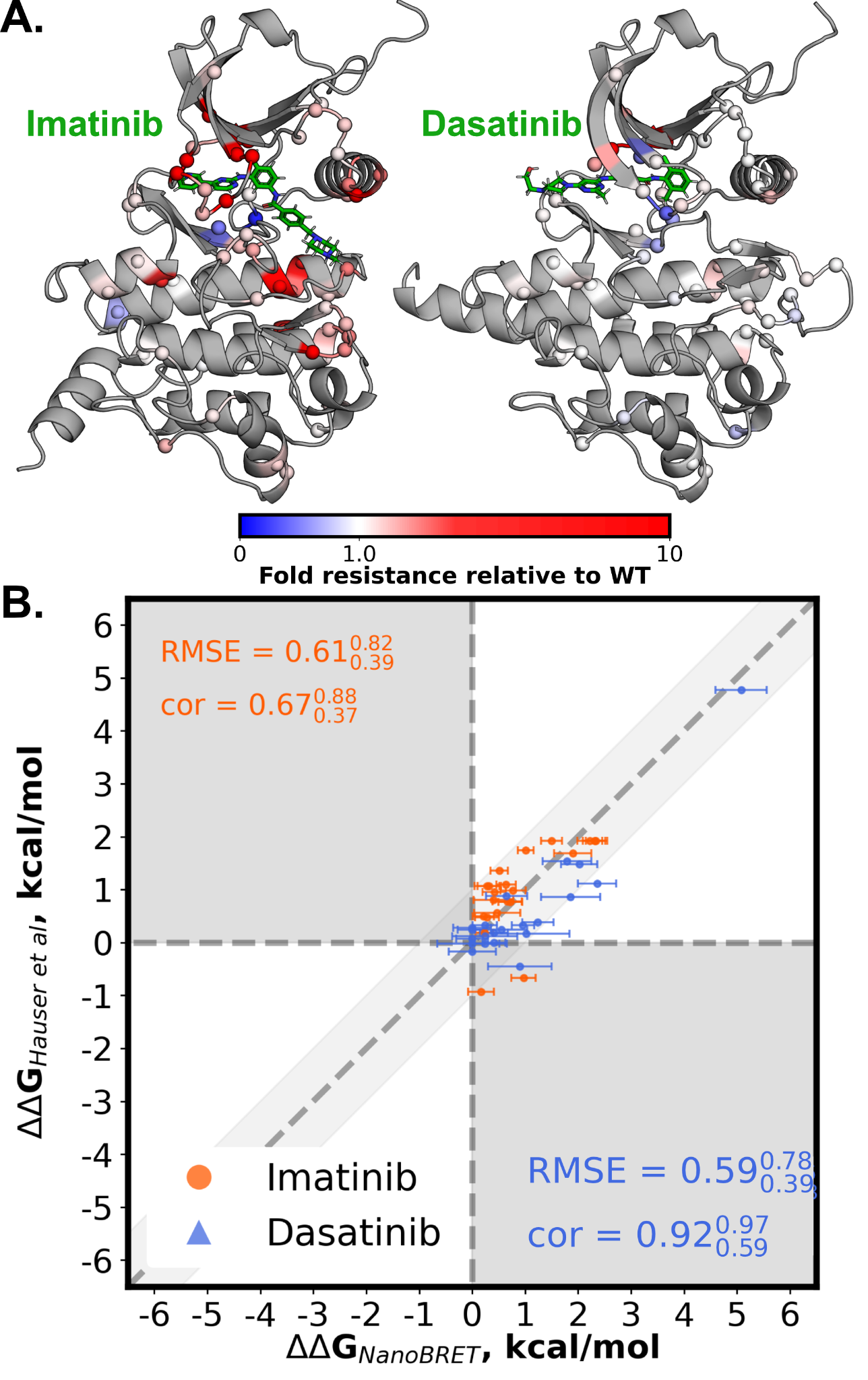
论文使用NanoBRET亲和力测定法,这是一种基于**生物发光共振能量转移(BRET)**的技术,用于在活体HEK293T细胞中测量药物与激酶靶点的结合。通过测量不同浓度的抑制剂(如伊马替尼和达沙替尼)对BRET比率的影响,计算出每种化合物/突变体对的表观亲和力。
结果与讨论
论文比较了NanoBRET结果与结构基础方法的预测能力,发现这些方法能够准确分类激酶突变为抑制剂耐药或敏感。物理模拟在估计远离激酶活性位点的突变的ΔΔG方面表现最佳。对于预测不佳的突变(如T315A和L298F),研究发现起始构象和质子化状态显著影响预测准确性。
结论
论文提供了一个用于估计突变对抑制剂结合亲和力影响的基准,强调了结构基础方法在识别肿瘤特异性突变的最佳疗法、预测临床数据缺乏情况下的耐药突变以及识别对已建立抑制剂的潜在敏化突变方面的潜在应用。
术语解释
- ΔΔG:结合自由能变化,衡量突变对抑制剂结合的影响。
- NanoBRET:一种用于测量分子间相互作用的技术,通过生物发光共振能量转移实现。
- PMX:用于分子动力学模拟中的自由能计算工具。
- flex_ddg:Rosetta中的一个协议,用于预测突变对蛋白质稳定性或结合亲和力的影响。
这篇论文通过结合实验和计算方法,为未来的研究提供了一个坚实的基础,帮助更好地理解和预测激酶突变对抑制剂结合的影响。
