IgDesign:使用逆折叠技术针对多种治疗性抗原进行体外验证的抗体设计
原标题:IgDesign: In vitro validated antibody design against multiple therapeutic antigens using inverse folding
5 分
关键词
摘要
深度学习方法已展示出在给定骨架结构的情况下设计蛋白质序列的能力[1, 2, 3, 4, 5]。虽然这些方法已在计算机模拟中应用于设计抗体互补决定区(CDRs),但尚未在体外验证其用于设计抗体结合物的效果,而这才是抗体设计成功的真正衡量标准。在此,我们描述了一种用于抗体CDR设计的深度学习方法IgDesign,并通过成功设计8种治疗性抗原的结合物展示了其稳健性。该模型的任务是使用抗体-抗原复合物的天然骨架结构以及抗原和抗体框架(FWR)序列作为背景,设计重链CDR3(HCDR3)或所有三个重链CDRs(HCDR123)。对于每种抗原,我们设计100个HCDR3和100个HCDR123,将它们嵌入到天然抗体的可变区中,并使用表面等离子共振(SPR)筛选它们与抗原的结合能力。作为基线,我们筛选了从模型训练集中提取的100个HCDR3,并与天然HCDR1和HCDR2配对。我们观察到,HCDR3设计和HCDR123设计均优于仅HCDR3的基线。IgDesign是第一个经过实验验证的抗体逆折叠模型。它能够以高成功率设计出针对多种治疗性抗原的抗体结合物,并在某些情况下表现出比临床验证的参考抗体更好的亲和力。抗体逆折叠在新型抗体设计和先导优化中都有应用,使IgDesign成为加速药物开发和实现治疗设计的宝贵工具。本研究生成的数据为多样化的抗体-抗原相互作用提供了有用的基准。我们使用这些数据来评估自洽性RMSD(scRMSD),并使用ABodyBuilder2 [6]、ABodyBuilder3 [7]和ESMFold [8]作为评估结合的指标。我们开源了IgDesign的代码和SPR数据集。代码和数据集可以在https://github.com/AbSciBio/igdesign找到。
AI理解论文
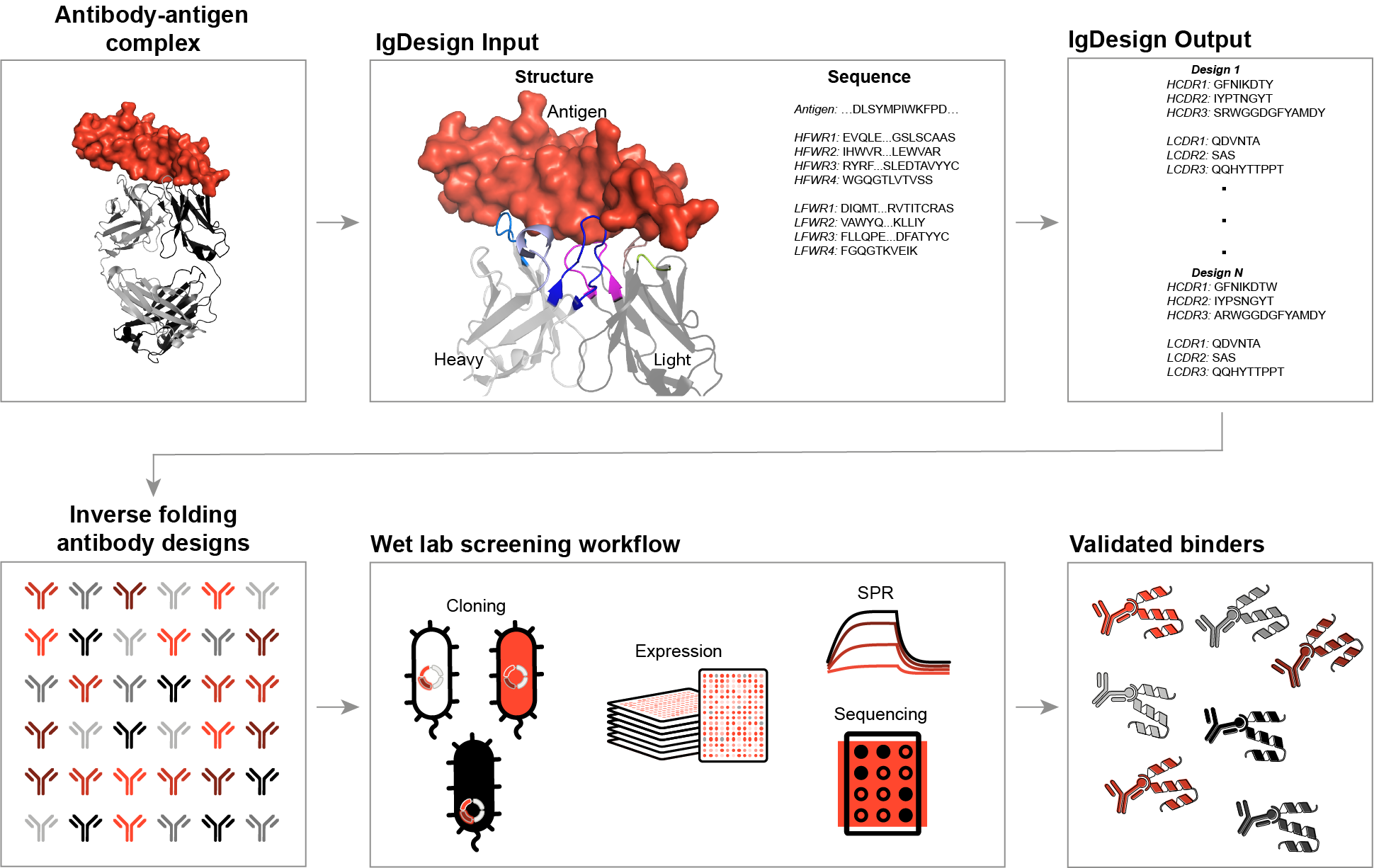
该文档主要探讨了抗体序列设计和**表面等离子体共振(SPR)**实验的结合应用,旨在通过机器学习模型优化抗体的设计和验证过程。以下是对文档的详细总结:
1. 研究背景与目标
文档的研究背景涉及抗体设计,这是生物制药领域的重要课题。传统的抗体设计方法通常依赖于实验筛选,耗时且成本高昂。本文的目标是通过机器学习模型,特别是IgDesign,来提高抗体设计的效率和准确性。IgDesign通过学习抗体-抗原复合物的结构和序列信息,生成具有高结合能力的抗体序列。
2. 方法与技术
2.1 IgDesign模型
IgDesign模型是基于深度学习的抗体设计工具。它通过输入抗体-抗原复合物的三维结构和序列信息,生成新的抗体序列。模型的训练数据来自SAbDab数据库,包含大量已知的抗体-抗原结构。IgDesign的设计流程包括以下步骤:
- 序列生成顺序:模型按照HCDR3、HCDR1、HCDR2、LCDR1、LCDR2、LCDR3的顺序生成序列,以避免在预测HCDR3时依赖可能错误的CDR预测。
- 采样方法:使用加权随机采样,并对模型的logits应用softmax函数,温度设为T=0.5。
2.2 表面等离子体共振(SPR)实验
SPR是一种用于检测分子间相互作用的技术。本文中,SPR用于验证IgDesign生成的抗体序列的结合能力。实验流程包括:
- 样品制备:通过裂解缓冲液处理样品,并使用高通量SPR设备进行检测。
- 实验设置:使用Carterra LSA SPR仪器,进行非再生动力学方法的抗原结合实验。
- 数据分析:通过传感图(sensorgrams)展示不同抗体与抗原的结合情况。
3. 实验结果与分析
实验结果表明,IgDesign生成的抗体序列在结合能力上显著优于基线模型。基线模型通过从训练集中采样HCDR3序列来进行比较,其成功率在大多数抗原上为0%。IgDesign的成功率则显著提高,表明模型能够有效利用输入特征进行外推。
4. 讨论与贡献
本文的主要贡献在于展示了IgDesign模型在抗体设计中的有效性,并通过SPR实验验证了其生成序列的功能性。研究表明,IgDesign不仅能够生成具有高结合能力的抗体序列,还能通过学习输入特征进行有效的序列设计。这为抗体药物的开发提供了一种高效且低成本的替代方案。
5. 结论
本文通过结合机器学习和SPR实验,提出了一种创新的抗体设计方法。IgDesign模型展示了其在抗体序列生成中的潜力,特别是在提高结合能力和设计效率方面。未来的研究可以进一步优化模型的输入特征和训练数据,以提高其在不同抗原上的适用性。
专业术语解释
- 抗体(Antibody):一种由免疫系统产生的蛋白质,用于识别和中和外来物质如细菌和病毒。
- 抗原(Antigen):能够引发免疫反应的物质,通常是病原体的一部分。
- CDR(Complementarity-Determining Region):抗体的互补决定区,是抗体与抗原结合的关键区域。
- SPR(Surface Plasmon Resonance):一种用于研究分子间相互作用的光学技术,通过检测表面等离子体波的变化来分析结合事件。
通过本文的研究,展示了机器学习在生物医药领域的应用潜力,尤其是在抗体设计和验证方面的创新应用。
