将结核病基因组学引入临床:一个全面的流程,用于从基因组数据预测抗菌药物敏感性,并经过ISO标准的验证和认证。
原标题:Bringing TB genomics to the clinic: A comprehensive pipeline to predict antimicrobial susceptibility from genomic data, validated and accredited to ISO standards
5 分
关键词
摘要
背景 全基因组测序(WGS)在结核病的临床管理中发挥着越来越重要的作用,但目前缺乏适合临床报告并符合国际标准认证的便捷生物信息学工具。方法 我们开发了tbtAMR,这是一种灵活而全面的工具,用于分析结核分枝杆菌的基因组数据,包括推断表型耐药性和谱系分类。通过使用本地和公开可用的真实世界数据(表型和基因型)以及合成基因组数据进行验证,以确定适当的质量控制指标,并广泛验证该流程在临床中的应用。结果 tbtAMR准确预测了第一线和第二线药物的谱系和表型耐药性,其计算和预测性能与当前可用的其他生物信息学工具相当或更好。tbtAMR具有灵活性,可以通过修改标准来满足用户的需求。解释 tbtAMR工具适用于临床和公共卫生微生物实验室环境,并且可以由非程序员根据特定的本地需求进行定制。我们已在实验室中根据ISO标准对该工具进行了认证,并已在临床相关时间框架内实施了基于基因组序列数据的AMR常规报告。提供了报告模板、验证方法和数据集,为实验室采用并寻求其自身认证这一关键测试提供了一条途径,以改善全球结核病的管理。资金来源 维多利亚州政府卫生部;澳大利亚国家健康和医学研究委员会及医学研究未来基金。研究背景 在本研究之前的证据 我们在PubMed上搜索了使用以下搜索词的研究:“结核分枝杆菌”、“临床”、“生物信息学”、“基因组学”、“药物耐药性(或抗菌)”,截至2024年7月31日发表的研究,不限语言(n=258)。我们考虑了所有使用基因组学推断结核分枝杆菌可能的药物耐药性的研究。这些研究中的许多都强调了从基因组数据中进行有意义解释以推断临床应用AMR的挑战。尽管开发了生物信息学工具并编制了耐药性突变目录,但这些研究中很少有直接解决实施全基因组测序(WGS)和生物信息学流程以提供经过验证和认证的临床应用结果的挑战,也没有提供可持续的解决方案。本研究的附加价值 据我们所知,本研究首次详细介绍了验证和认证从WGS数据中常规推断AMR用于结核分枝杆菌治疗的临床应用的实际解决方案。我们开发、验证并认证了一种生物信息学工具tbtAMR,该工具稳健且灵活,采用数据驱动的方法推断耐药性,允许在大量内部数据和公开可用数据的基础上简单定制行为。此外,我们已将此流程和相关数据提供给更广泛的公共卫生社区,以帮助其他人实施类似的程序。与传染病临床医生、微生物学家和流行病学家的咨询使我们能够开发出一个稳健的工作流程,涵盖测序、分析、解释、报告、差异解决和持续验证。所有可用证据的意义 本研究解决了在临床和公共卫生实验室中报告结核分枝杆菌基因组AMR的具体需求。此外,这项工作提供了一个流程和数据供实验室利用,以实施结核分枝杆菌的基因组AMR。通过使用和进一步开发公开可用和开源的生物信息学软件,我们证明了在低发病率高收入环境中临床报告结核分枝杆菌基因组AMR结果的可行性。我们相信,这项工作为使结核分枝杆菌基因组AMR的报告对公共卫生和参考实验室更具可访问性奠定了基础。
AI理解论文
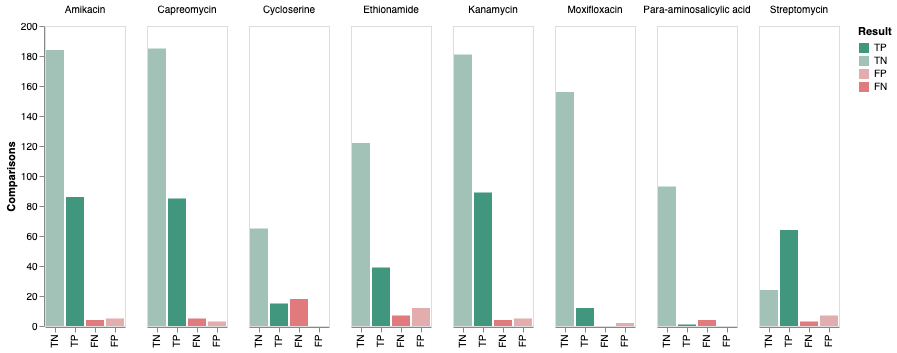


这篇论文的主要内容是介绍一种名为tbtAMR的综合管道,该管道用于从基因组数据中预测结核分枝杆菌(Mycobacterium tuberculosis,简称Mtb)的抗菌药物敏感性,并已通过ISO标准的验证和认证。以下是对论文的详细总结:
背景与动机
论文指出,**结核病(TB)**是由结核分枝杆菌引起的疾病,主要影响低收入和中等收入社区,是单一传染病导致死亡的主要原因。治疗结核病的过程漫长且昂贵,通常需要至少六个月的多药联合治疗。**全基因组测序(WGS)**显示出改善结核病临床管理的潜力,但目前缺乏适合临床报告并符合ISO标准的生物信息学工具。
方法
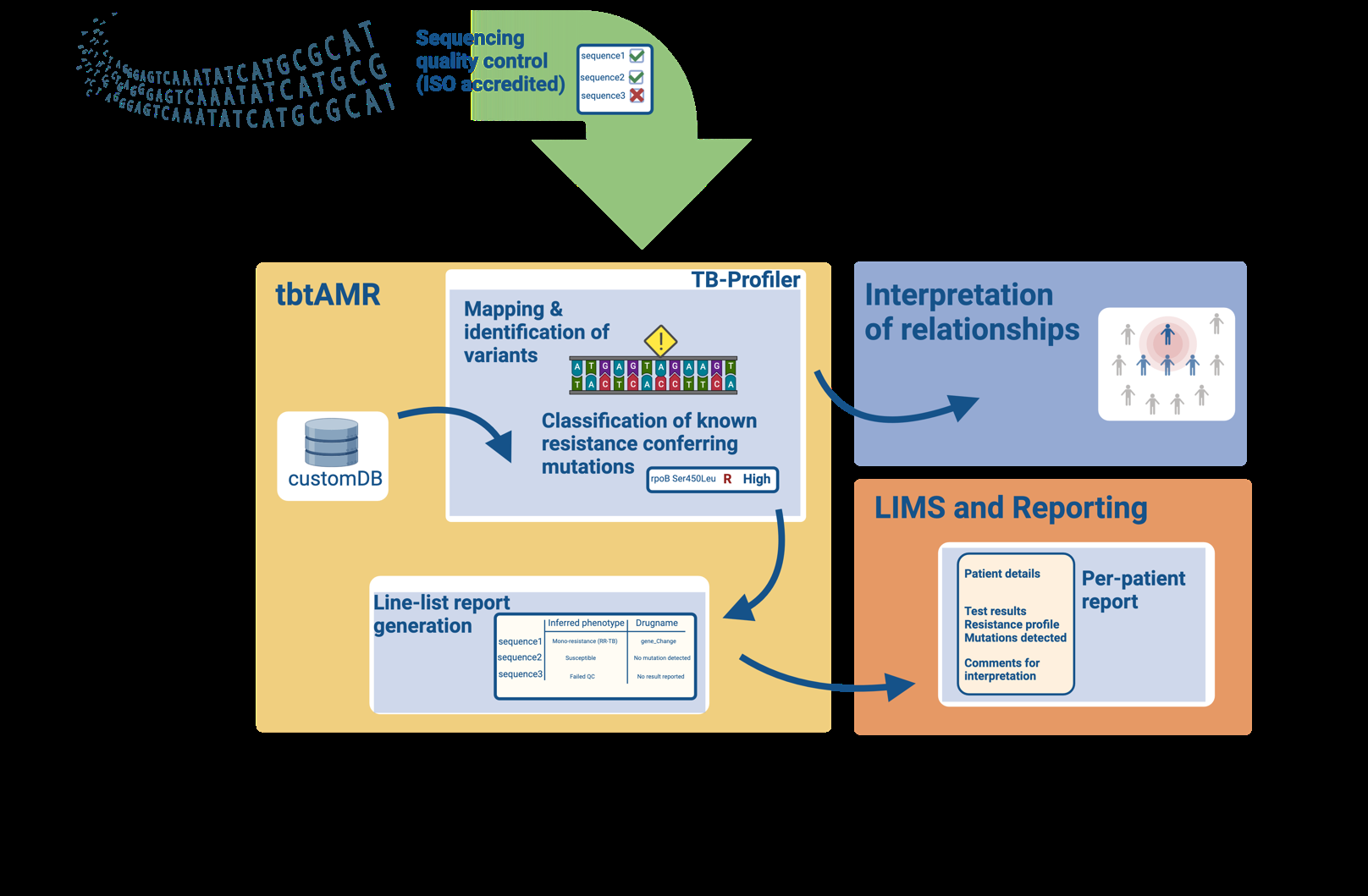
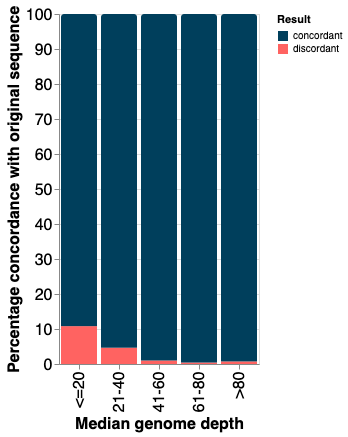
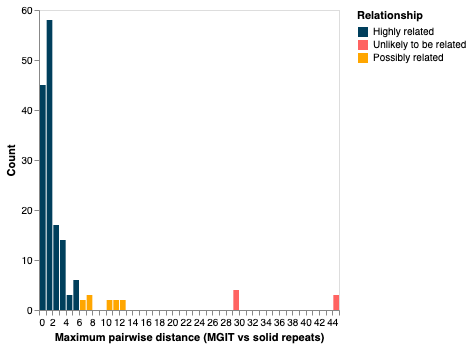
研究团队开发了tbtAMR,这是一个用于分析结核分枝杆菌基因组数据的综合管道。该管道包括从固体和液体培养(如MGIT培养)中推断表型敏感性和谱系分类。研究人员使用本地和公开的真实世界数据(包括表型和基因型数据)以及合成基因组数据来确定适当的质量控制指标,并对该管道进行了广泛的临床使用验证。
- MGIT培养:指的是使用Mycobacterial Growth Indicator Tubes进行的液体培养方法。
- 表型敏感性:指的是细菌对特定抗菌药物的反应性。
- 谱系分类:指的是根据基因组数据对结核分枝杆菌进行分类。
研究发现
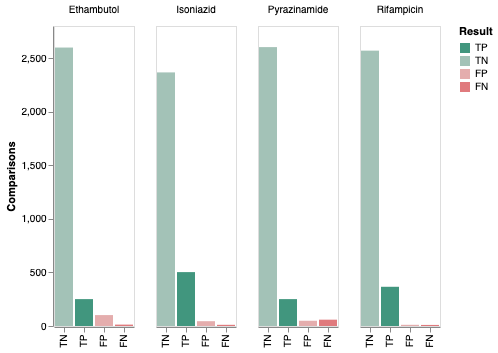
tbtAMR能够准确预测结核分枝杆菌的谱系和对一线和二线药物的表型敏感性,包括从MGIT培养中获得的数据。研究团队设计并实施了一种适合临床和公共卫生用户的报告模板,并将该管道认证为符合ISO标准。
解释与贡献
- 准确性:tbtAMR在预测谱系和表型敏感性方面表现出高准确性,特别是在处理MGIT培养数据时。
- 报告模板:提供了一种适合临床和公共卫生使用的报告模板,帮助实验室采用并寻求对这一关键测试的认证,以改善全球结核病管理。
- 验证与质量控制:通过使用真实世界数据和合成数据,研究团队对tbtAMR进行了广泛的验证,确保其在临床应用中的可靠性。
结论
tbtAMR管道被证明是准确且适合用于临床和公共卫生的工具。研究提供了报告模板、验证方法和数据集,为实验室采用这一工具并寻求认证提供了路径。该研究未获得特定资金支持。
术语解释
- ISO标准:国际标准化组织制定的标准,确保产品、服务和系统的质量、安全性和效率。
- 生物信息学工具:用于分析生物数据的软件工具,特别是在基因组学领域。
- 抗菌药物敏感性:指细菌对抗菌药物的反应性,决定了药物的有效性。
研究的局限性与未来方向
尽管tbtAMR在预测抗药性方面表现出色,但对于某些药物(如bedaquiline和linezolid)的抗药性预测仍存在挑战。未来的研究可能需要进一步完善这些药物的基因组预测能力。
通过这篇论文,研究团队为结核病的基因组学研究提供了一个重要的工具,推动了结核病的临床管理和公共卫生策略的进步。
