用于探索酿酒酵母和裂殖酵母的酵母知识图谱数据库
原标题:Yeast Knowledge Graphs Database for Exploring Saccharomyces cerevisiae and Schizosaccharomyces pombe
5 分
关键词
摘要
生物医学文献中包含了关于基因和蛋白质功能在各种生物过程和疾病中的大量信息。然而,导航这些庞大且通常受限的数据可能具有挑战性,使得高效提取特定见解变得困难。在这项研究中,我们介绍了一种高通量流程,该流程利用OpenAI的生成预训练变换模型(GPT)来自动提取和分析基因功能信息。我们将这种方法应用于84,427篇关于酿酒酵母的出版物和6,452篇关于裂殖酵母的出版物,识别出3,432,749个关于出芽酵母的关系和421,198个关于裂殖酵母的关系。这产生了一个全面的、可搜索的在线知识图数据库,可在yeast.connectome.tools和spombe.connectome.tools访问,为用户提供对各种相互作用和路径的广泛访问。我们的分析强调了将人工智能与生物信息学相结合的强大能力,通过对Hsp104和Atg8蛋白等重要节点的关键见解得以证明。这项工作不仅促进了酵母研究中的高效数据提取,还为其他生物系统中的类似研究提供了一个可扩展的模型。亮点:从全文研究文章中生成酵母知识图谱。分析了超过90,000篇关于酿酒酵母和裂殖酵母物种的出版物。使用基于GPT的自然语言处理提取了数百万个关系。通过交互式网络平台和API访问酵母知识图谱。先进工具使得对基因网络和功能相互作用的见解成为可能。
AI理解论文


该文档介绍了一项利用生成式预训练变换器(GPT)模型进行自然语言处理的创新研究,旨在从大量科学文献中提取酵母基因功能信息。研究的核心是一个由OpenAI的GPT-3.5 turbo模型驱动的复杂文本挖掘管道。以下是该文档的主要内容:
研究背景与动机
背景:传统上,研究人员依赖手动整理的数据库来组织和访问经过实验验证的“黄金标准”数据。然而,这些数据库面临数据整理延迟的问题,无法跟上新信息的涌入。这种延迟限制了研究人员实时理解酵母生物学复杂交互的能力。
动机:为了弥补这一差距,研究团队引入了一种自动化的高通量系统,利用GPT模型的自然语言处理能力来加速新发表数据的整合。
方法
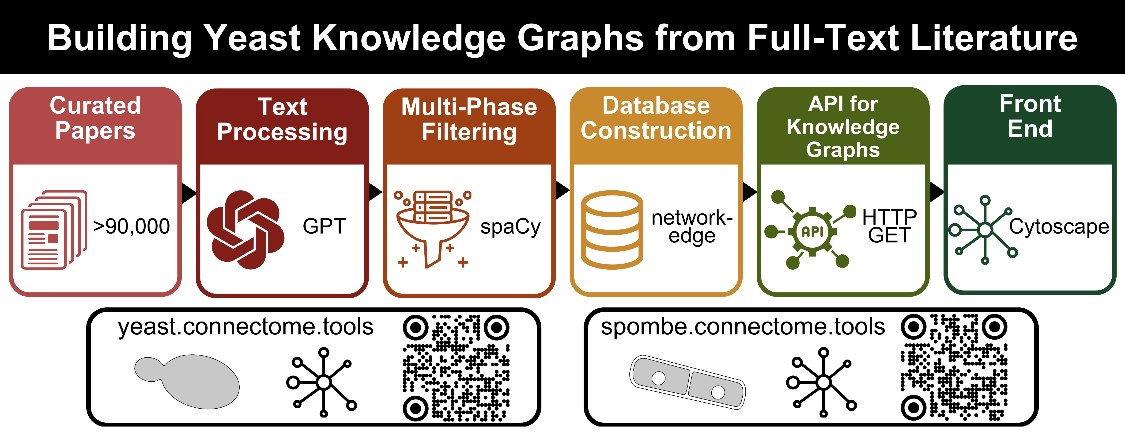
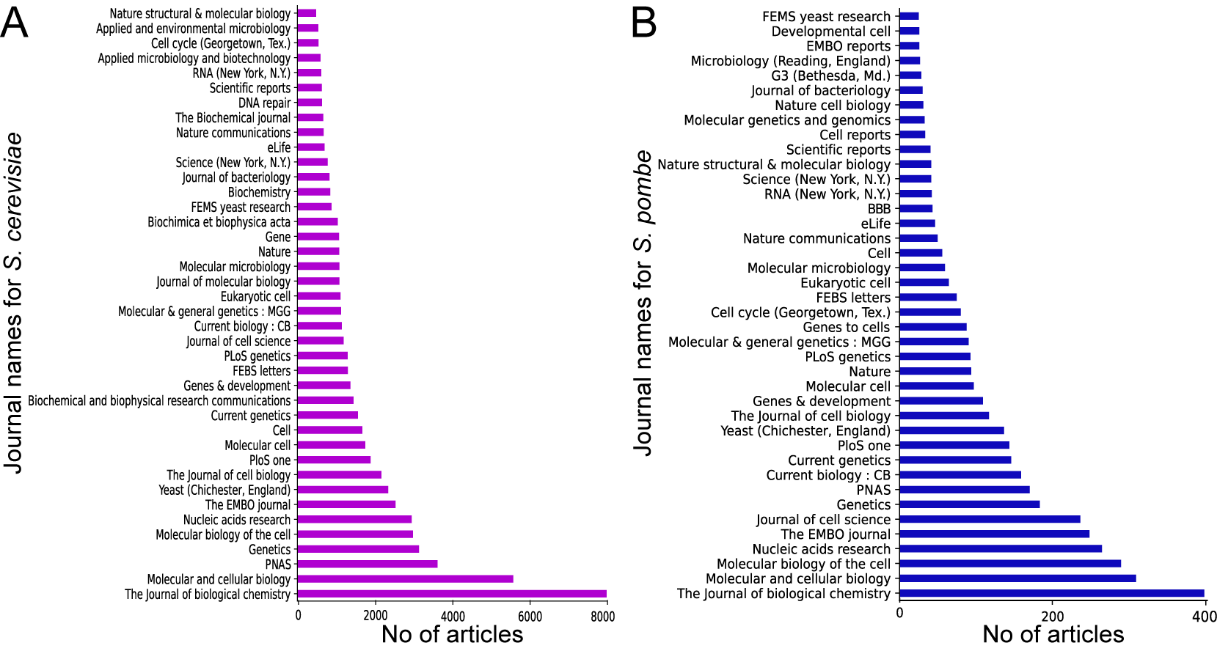
文献检索与预处理:研究团队从YeastMine数据库和UniProt中获取了酵母基因及其别名的广泛列表,并使用这些列表创建了专门的搜索查询,以在PubMed中定位相关研究文章。使用Bio.Entrez包自动化检索文章,并通过Elsevier API获取全文,补充数据集。
文本处理:使用Python脚本和OpenAI的GPT-3.5-turbo模型处理文本,提取基因特定信息。模型配置为零温度设置,以优先考虑准确性,减少随机性。设计了定制的提示结构输出,确保每行突出显示实体对及其特定关系。
多阶段过滤与数据库构建:初始生成的关系边经过多阶段过滤过程进行验证和增强,使用spaCy NLP库验证生成的边,排除任何虚构的边。最终的边用于构建网络边数据库,使用Python-Flask框架和Networkx进行图分析,MongoDB进行数据库管理。
结果
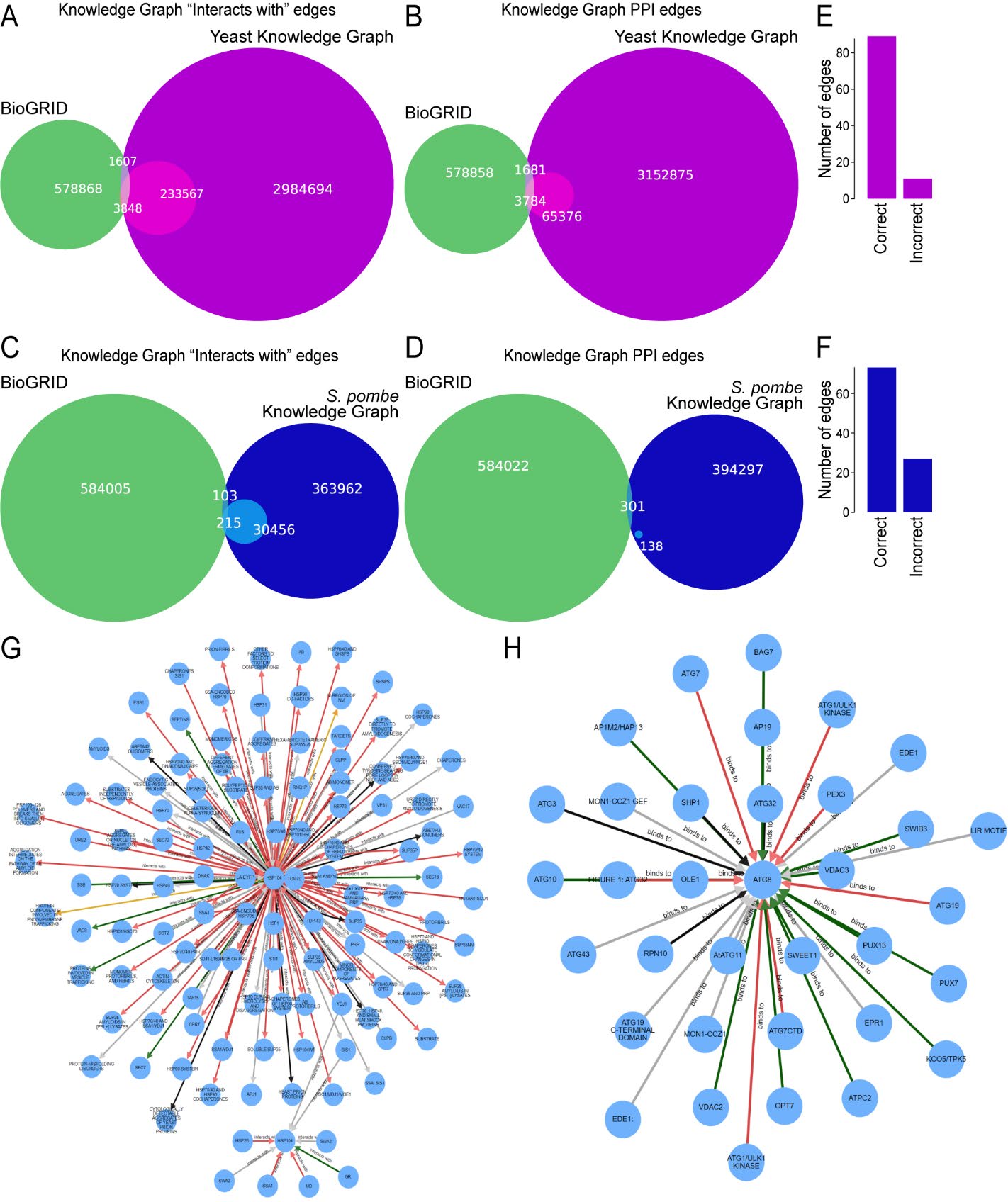
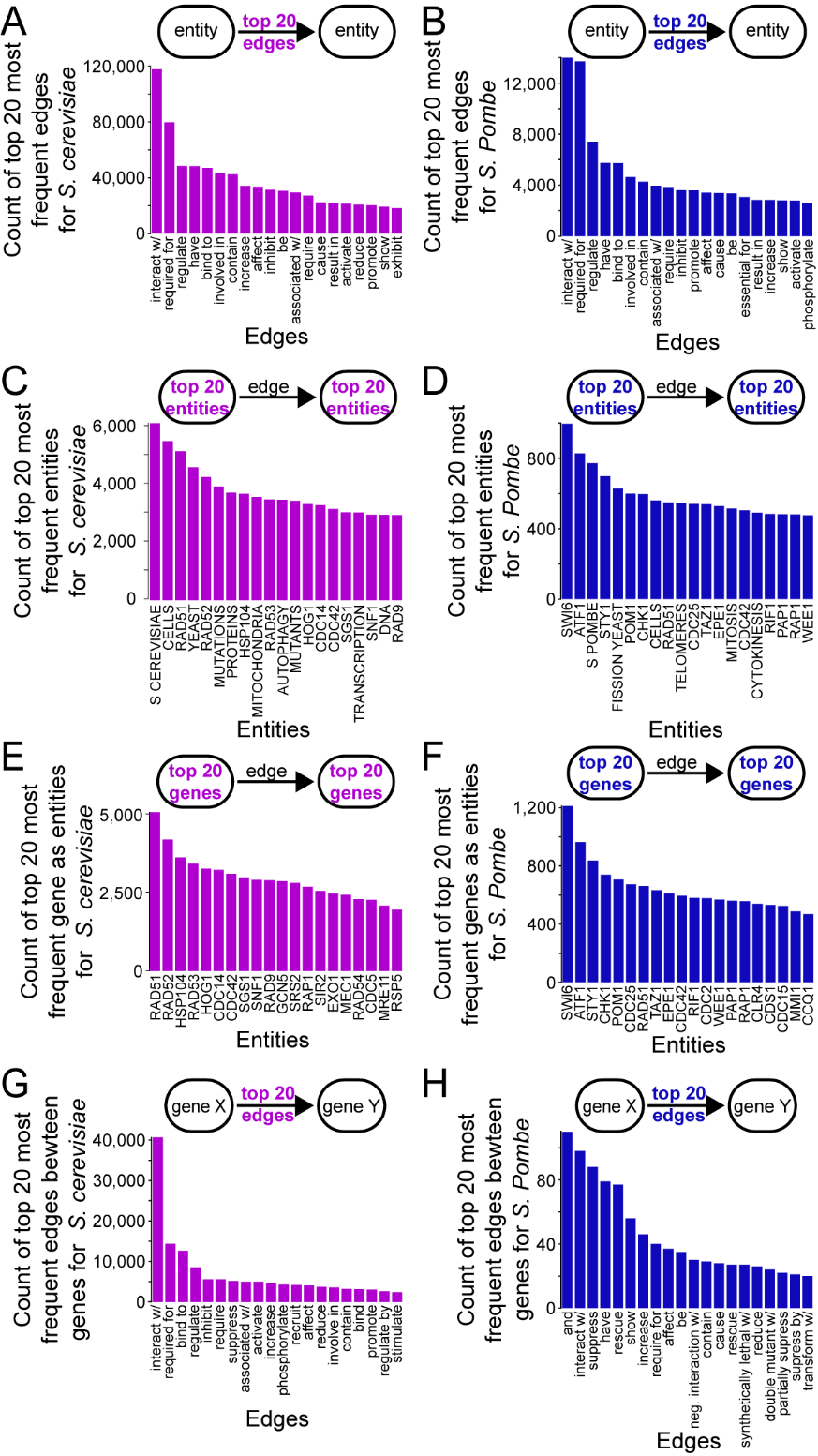
知识图谱构建:使用GPT-3.5模型构建了酵母知识图谱(YeastKnowledgeGraph)和裂殖酵母知识图谱(FissionYeastKnowledgeGraph)。处理了84,427篇关于S. cerevisiae的出版物和6,452篇关于S. pombe的出版物,分别创建了3,432,749和421,198个关系。
交互特性与用户参与:知识网络可视化提供了动态交互体验,用户可以直接与网络互动,选择节点以激活工具提示,提供进一步探索的选项。网络高度可定制,用户可以删除节点或集群,按特定关系类型过滤,并调整布局。
与BioGRID的比较:与BioGRID的蛋白质-蛋白质相互作用(PPI)网络进行比较,YeastKnowledgeGraph识别了233,567个BioGRID中不存在的独特交互边,展示了其增强的检测能力。
讨论
优势:该研究展示了生成式AI模型在推进生物研究中的潜力,知识图谱提供了理解酵母基因交互和生物途径的宝贵资源。
局限性:依赖于公开可用的文献和机构订阅的文章,可能导致最新研究结果的整合延迟。此外,自动化技术在精确性上与手动整理相比存在权衡。
结论
该研究通过创建酵母知识图谱和裂殖酵母知识图谱,展示了生成式AI模型在生物研究中的应用潜力。这些知识图谱通过交互式网络平台提供访问,为理解酵母中的基因交互和生物途径提供了宝贵资源。研究为进一步研究复杂生物系统奠定了基础,具有扩展到其他模式生物的潜力。
术语解释
- 生成式预训练变换器(GPT):一种深度学习模型,擅长自然语言处理任务,通过大量文本数据进行预训练。
- 知识图谱:一种数据结构,表示实体及其相互关系,常用于组织和分析复杂信息。
- 高通量系统:一种能够快速处理大量数据的系统,通常用于加速科学研究中的数据分析。
通过这项研究,研究团队展示了如何利用先进的自然语言处理技术来加速生物信息学领域的数据整合和知识发现。
